Генерация и отбор признаков
В реальных задачах вы столкнетесь с широким спектром трудностей, и одним из первых этапов будет подготовка данных. Уже на этом этапе есть ряд возможных проблем, например:
Нехватка данных — у вас может быть мало данных. Более того, понять, какой объем данных необходим — тоже отдельная задача. Другая вариация похожей проблемы — это большой объем данных, но без разметки.
Некачественная разметка — даже в широко известных MNIST, CIFAR-10 и ImageNet есть ошибки в разметке 🎮[demo]. В вашем датасете они тоже будут. Важно понимать, что разметка может не являться бесспорным эталоном.
Шум в данных — полезно подумать о потенциальной зашумленности данных. Например, когда люди заполняют таблицы, они могут допускать ошибки. Хорошо понимать, какого рода ошибки могут содержаться в ваших данных.
Несбалансированность датасета — какие-то классы могут быть плохо представлены (минорные классы). Например, если в вашем датасете будет всего 10 фотографий одного класса и 10 000 другого, то модели будет очень заманчиво вообще не пытаться определять минорный класс (всего 0.1% ошибок).
Ковариантный сдвиг — явление, когда признаки тренировочной и тестовой выборок распределены по-разному. Из-за этого мы можем получать плохие предсказания на тестовой выборке, когда модель просто не знает, что признаки могут находиться в другом распределении.
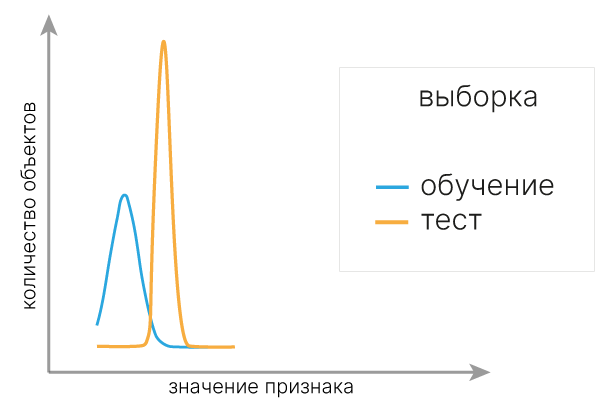
Практический совет: для быстрого обнаружения ковариантного сдвига можно обучить модель, которая будет предсказывать, относится ли объект к train или test выборке. Если модель легко делит данные по такому принципу, то имеет смысл визуализировать значения признаков, по которым она это делает.
Малое число источников данных — проблема, родственная предыдущей. В вашем датасете могут быть данные только от одного прибора или одной модели прибора. Могут быть данные, снятые только одним специалистом, или в одной больнице, или только у взрослых. Это также может влиять на способность вашего алгоритма обобщать полученное решение и требует пристального внимания.
Грязные данные — в данных могут быть полные/неполные дубликаты, пропуски, ошибки форматов, перемешанные столбцы и многое другое. Такие проблемы могут быть как естественной характеристикой (нет данных о каких-то объектах, отсюда пропуски), так и ошибкой при агрегации данных из разных источников. Важно подумать о проверках и тестах, чтобы быть уверенным в качестве.
Все это приводит к целому спектру сложностей, из которых самой типичной будет переобучение модели — какую бы простую модель вы не взяли, она все равно будет выучивать искажения вашего датасета.
Прежде всего надо убедиться, что датасет сбалансирован:
import torch
import matplotlib.pyplot as plt
from sklearn.datasets import load_wine
def show_class_balance(y, classes):
_, counts = torch.unique(torch.tensor(y), return_counts=True)
plt.bar(classes, counts)
plt.ylabel("n_samples")
plt.ylim([0, 75])
plt.show()
wine = load_wine()
classes = wine.target_names
show_class_balance(wine.target, classes)
Разница в 10–20% будет незначительна, поэтому для наглядности мы искусственно разбалансируем наш датасет при помощи метода make_imbalance 🛠️[doc] из библиотеки imbalanced-learn 🛠️[doc].
from imblearn.datasets import make_imbalance
x, y = make_imbalance(
wine.data, wine.target, sampling_strategy={0: 10, 1: 70, 2: 40}, random_state=42
)
show_class_balance(y, classes)
Если в данных нехватка именно конкретного класса, то можно бороться с этим при помощи разных способов сэмплирования.
Важно понимать, что в большинстве случаев данные, полученные таким способом, должны использоваться в качестве обучающего набора, но ни в коем случае не в качестве валидации или теста.
Мы можем увеличить число объектов меньшего класса за счет дублирования.
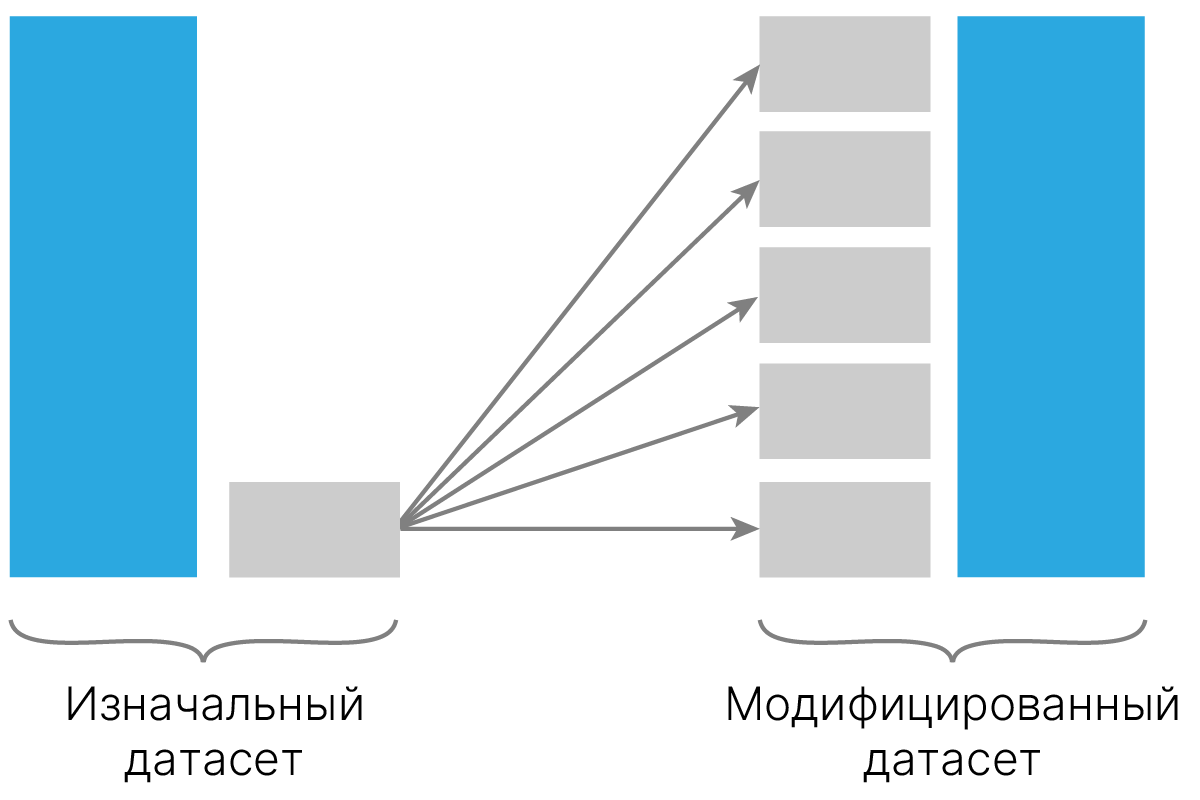
Такой oversampling может быть выполнен с помощью класса RandomOverSampler 🛠️[doc] из imbalanced-learn, как показано ниже:
from imblearn.over_sampling import RandomOverSampler
ros = RandomOverSampler(random_state=0)
x_ros, y_ros = ros.fit_resample(x, y)
show_class_balance(y_ros, classes)
Аналогично, можно взять для обучения не всех представителей большего класса.
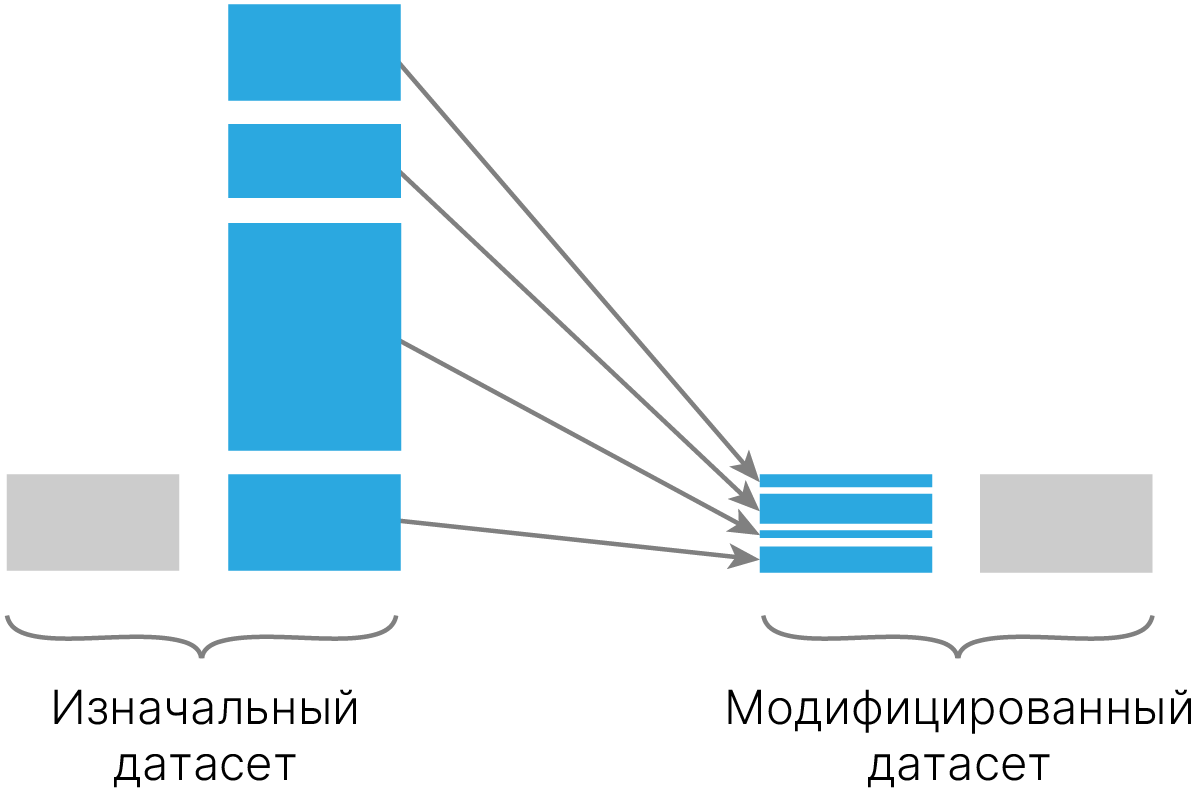
from imblearn.under_sampling import RandomUnderSampler
rus = RandomUnderSampler(random_state=42)
x_res, y_res = rus.fit_resample(x, y)
show_class_balance(y_res, classes)
Минус подхода: мы можем выбросить важных представителей большего класса, ответственных за существенное улучшение генерализации, и из-за этого качество модели существенно ухудшится.
Выбрасывать объекты большего класса можно разными способами. Например, кластеризовать объекты большего класса и брать по заданному количеству объектов из каждого класса.
Можно использовать ансамбли вместе с undersampling. В этом случае мы можем, к примеру, делать сэмплирование только большего класса, а объекты минорного класса оставлять как есть.
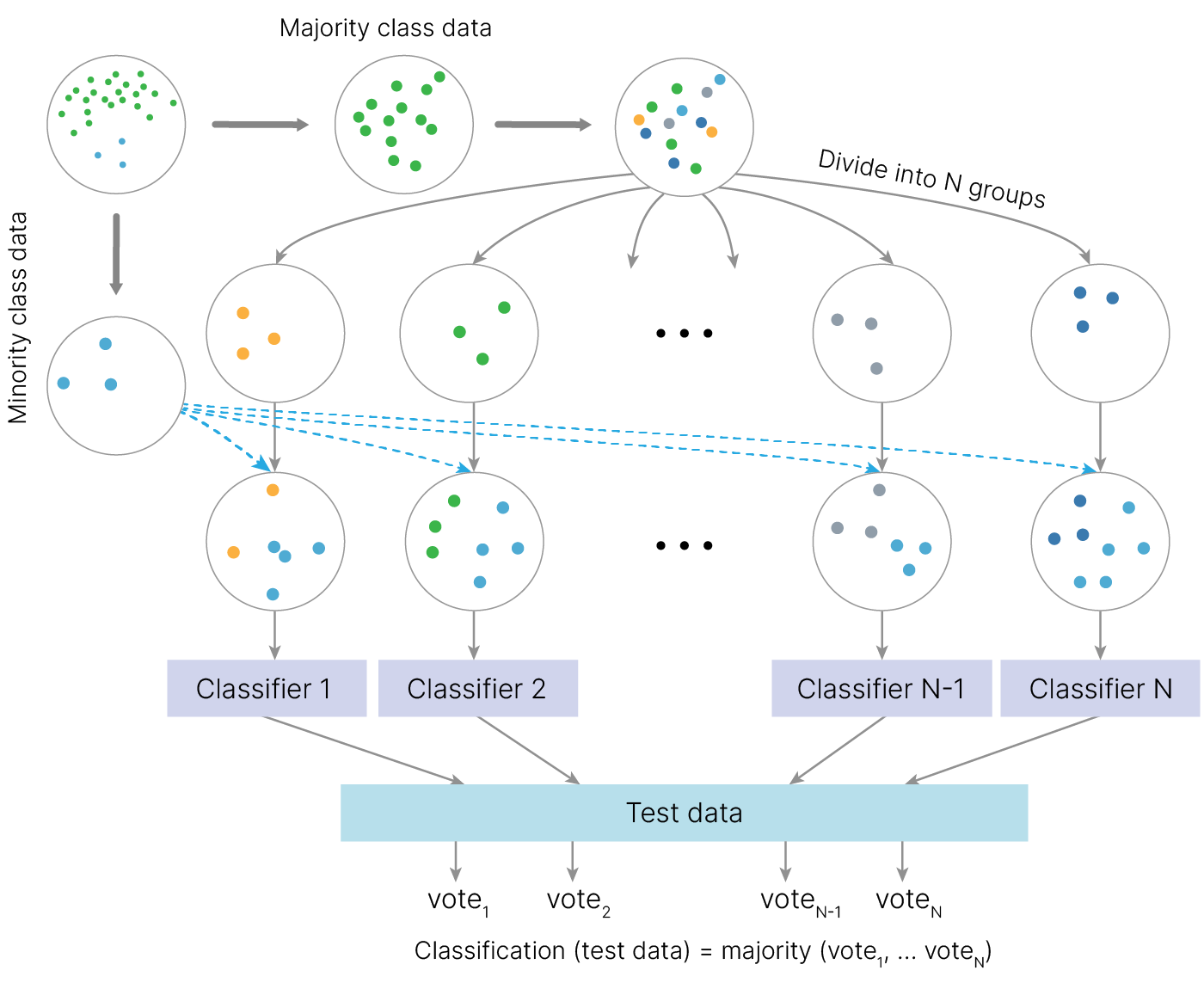
Или просто сэмплировать объекты и того, и другого класса в равном количестве.
Другой подход к решению этой проблемы — создание синтетических данных.
Synthetic Minority Over-sampling Technique (SMOTE) позволяет генерировать синтетические данные за счет реальных объектов из минорного класса.
Алгоритм работает следующим образом:
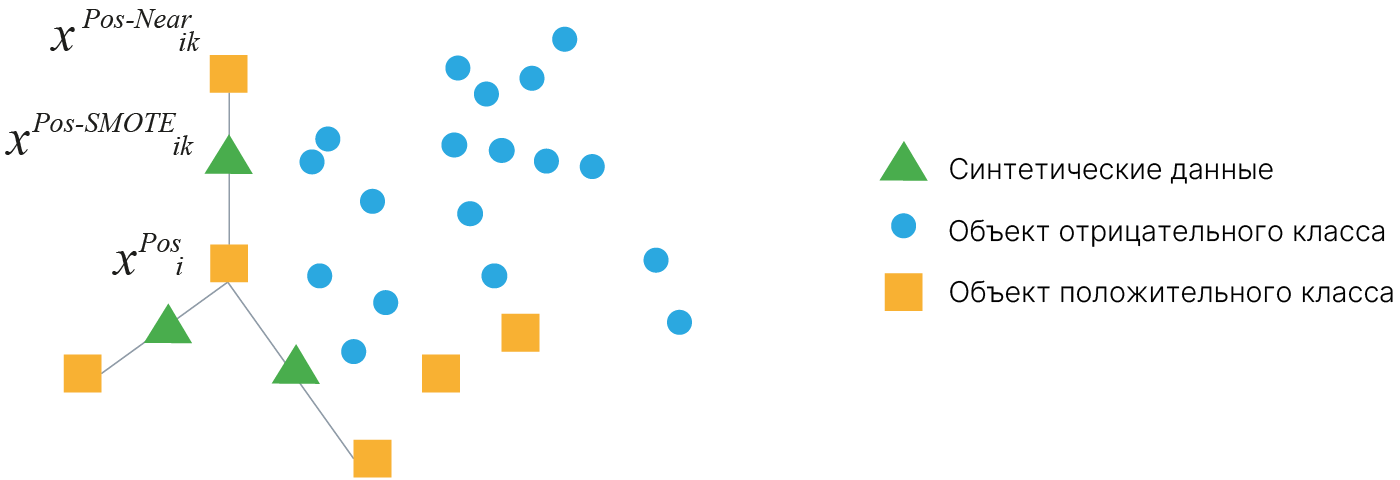
Число соседей, как и число раз, которое мы запускаем описанную выше процедуру, можно регулировать.
from imblearn.over_sampling import SMOTE
oversample = SMOTE()
x_smote, y_smote = oversample.fit_resample(x, y)
show_class_balance(y_smote, classes)
Количество объектов каждого класса, которое должно получиться после генерации, можно задать явно:
over = SMOTE(sampling_strategy={0: 20, 1: 70, 2: 70})
x_smote, y_smote = over.fit_resample(x, y)
show_class_balance(y_smote, classes)
[blog] ✏️ SMOTE for Imbalanced Classification with Python — подробнее про использование пакета.
Обращайте внимание на то, какие метрики вы используете. При решении задачи классификации часто используется accuracy (точность), равная доле правильно классифицированных объектов. Эта метрика позволяет адекватно оценить результат классификации в случае сбалансированных классов. В случае дисбаланса классов данная метрика может выдать обманчиво хороший результат.
Пример: датасет, в котором 95% объектов относятся к классу 0 и 5% к классу 1.
from sklearn.datasets import make_classification
from collections import Counter
import numpy as np
import matplotlib.pyplot as plt
x, y = make_classification(
n_samples=1000,
n_features=2,
n_redundant=0,
n_clusters_per_class=1,
weights=[0.95],
flip_y=0,
random_state=42,
)
counter = Counter(y)
print("Class distribution ", Counter(y))
for label, _ in counter.items():
row_ix = np.where(y == label)[0]
plt.scatter(x[row_ix, 0], x[row_ix, 1], label=str(label))
plt.legend()
plt.show()
Class distribution Counter({0: 950, 1: 50})
И модель, которая для всех данных выдает класс 0:
class DummyModel:
def predict(self, x):
return np.zeros(x.shape[0]) # always predict class 0
Такая модель будет иметь $\text{accuracy} = 0.95$, хотя не выдает никакой полезной информации:
from sklearn.metrics import accuracy_score
dummy_model = DummyModel()
y_pred = dummy_model.predict(x)
accuracy = accuracy_score(y, y_pred)
print("Accuracy", accuracy)
Accuracy 0.95
Для несбалансированных данных лучше выбирать F1 Score 🛠️[doc], MCC 🛠️[doc] (Matthews correlation coefficient, коэффициент корреляции Мэтьюса) или balanced accuracy 🛠️[doc] (среднее между recall разных классов).
from sklearn.metrics import f1_score, matthews_corrcoef, balanced_accuracy_score
print("F1", f1_score(y, y_pred))
print("MCC", matthews_corrcoef(y, y_pred))
print("Balanced accuracy", balanced_accuracy_score(y, y_pred))
F1 0.0 MCC 0.0 Balanced accuracy 0.5
Эти метрики более адекватно оценивают качество модели при наличии дисбаланса классов, и в данном случае отражают отсутствие связи предсказаний с данными на входе модели.
В случае сильно несбалансированных наборов данных стоит задуматься, могут ли такие примеры рассматриваться как аномалия (выброс) или нет. Если такое событие и впрямь может считаться аномальным, мы можем использовать такие модели, как OneClassSVM, методы кластеризации или методы обнаружения гауссовских аномалий.
Эти методы требуют изменения взгляда на задачу: мы будем рассматривать аномалии как отдельный класс выбросов. Это может помочь нам найти новые способы разделения и классификации.
Пусть мы хотим отличать фрукты по каким-то признакам, скажем, по форме и цвету, и мы имеем выборку, где много яблок и груш. Задача обнаружения аномалий возникает, если мы предполагаем, что среди яблок и груш может вдруг возникнуть клубника и нам нужно отнести ее не к одному из известных классов, а выделить в отдельный, отличающийся класс.

Проблемой при работе с аномалиями является то, что аномальных значений может быть очень мало или вообще не быть. В таком случае алгоритм учит паттерны нормального поведения и реагирует на отличия от паттернов.
Разберем примеры обнаружения аномалий с помощью трех алгоритмов из библиотеки Scikit-Learn (там можно найти еще много различных алгоритмов).
Создадим датасет из двух кластеров и случайных значений.
import numpy as np
rng = np.random.RandomState(42)
# Train
x = 0.3 * rng.randn(100, 2) # 100 2D points
x_train = np.r_[x + 2, x - 2] # split into two clusters
# Test norlmal
x = 0.3 * rng.randn(20, 2) # 20 2D points
x_test_normal = np.r_[x + 2, x - 2] # split into two clusters
# Test outliers
x_test_outliers = rng.uniform(low=-4, high=4, size=(20, 2))
Напишем функцию визуализации, которая будет изображать созданный датасет на рисунке слева, а результат поиска аномалий — на рисунке справа.
def plot_outliers(x_train, x_test_normal, x_test_outliers, model=None):
fig, (plt_data, plt_model) = plt.subplots(1, 2, figsize=(12, 6))
plt_data.set_title("Created Dataset (real labels)")
plot_train = plt_data.scatter(
x_train[:, 0], x_train[:, 1], c="white", s=40, edgecolor="k"
)
plot_test_normal = plt_data.scatter(
x_test_normal[:, 0], x_test_normal[:, 1], c="green", s=40, edgecolor="k"
)
plot_test_outliers = plt_data.scatter(
x_test_outliers[:, 0], x_test_outliers[:, 1], c="red", s=40, edgecolor="k"
)
plt_data.set_xlim((-5, 5))
plt_data.set_ylim((-5, 5))
plt_data.legend(
[plot_train, plot_test_normal, plot_test_outliers],
["train", "test normal", "test outliers"],
loc="lower right",
)
if model:
plt_model.set_title("Model Results")
# plot decision function
xx, yy = np.meshgrid(np.linspace(-5, 5, 50), np.linspace(-5, 5, 50))
Z = model.decision_function(np.c_[xx.ravel(), yy.ravel()])
Z = Z.reshape(xx.shape)
plt_model.contourf(xx, yy, Z, cmap=plt.cm.Blues_r)
# plot prediction
full_data = np.concatenate((x_train, x_test_normal, x_test_outliers), axis=0)
predicted = model.predict(full_data)
anom_index = np.where(predicted == -1)
anom_values = full_data[anom_index]
plot_all_data = plt_model.scatter(
full_data[:, 0], full_data[:, 1], c="white", s=40, edgecolor="k"
)
plot_anom_data = plt_model.scatter(
anom_values[:, 0], anom_values[:, 1], c="red", s=40, marker="x"
)
plt_model.legend(
[plot_all_data, plot_anom_data],
["normal", "outliers"],
loc="lower right",
)
plt.show()
Посмотрим, как работает на этих данных алгоритм OneClassSVM 🛠️[doc].
Идея алгоритма состоит в поиске функции, которая положительна для областей с высокой плотностью и отрицательна для областей с малой плотностью.
Подробнее об алгоритме:
from sklearn.svm import OneClassSVM
gamma = 2.0 # Kernel coefficient
contamination = 0.05 # threshold
model = OneClassSVM(gamma=gamma, kernel="rbf", nu=contamination)
model.fit(x_train)
plot_outliers(x_train, x_test_normal, x_test_outliers, model)
Посмотрим, как на этих же данных работает алгоритм Isolation Forest 🛠️[doc].
Isolation Forest состоит из деревьев, которые «изолируют» (пытаются отделить от остальной выборки) наблюдения, случайным образом выбирая признак и случайное значение порога для этого признака (между max и min значениями признака). Такой алгоритм чаще и проще отделяет значения аномалий. Если построить по такому принципу множество деревьев, то значения, которые чаще других отделяются раньше, будут аномалиями.
На практике часто используют более продвинутую версию Extended Isolation Forest 🐾[git].
from sklearn.ensemble import IsolationForest
n_estimators = 200
contamination = 0.05 # threshold
model = IsolationForest(
n_estimators=n_estimators, contamination=contamination, random_state=rng
)
model.fit(x_train)
plot_outliers(x_train, x_test_normal, x_test_outliers, model)
Последним алгоритмом, на который мы посмотрим, будет Local Outlier Factor 🛠️[doc].
В нем используется метод k-NN. Расстояние до ближайших соседей используется для оценки расположения точек. Если соседи далеко, то точка с большой вероятностью является аномалией.
from sklearn.neighbors import LocalOutlierFactor
n_neighbors = 10
contamination = 0.05 # threshold
model = LocalOutlierFactor(
n_neighbors=n_neighbors, novelty=True, contamination=contamination
)
model.fit(x_train)
plot_outliers(x_train, x_test_normal, x_test_outliers, model)
Отдельное время стоит посвятить подбору порога для алгоритма.
Порог является граничным значением, выше которого данные считаются аномальными. Выбор порога — это компромиссное решение: слишком низкий порог может привести к ложным срабатываниям, тогда как слишком высокий порог может пропустить аномалии. Поэтому выбор порога напрямую связан с задачей и требованиям к детекции.
Построим матрицы ошибок (Confusion matrix) для разных порогов:
full_data = np.concatenate((x_train, x_test_normal, x_test_outliers), axis=0)
predicted = model.predict(full_data)
# change threshold to 0.005
model_2 = LocalOutlierFactor(n_neighbors=10, novelty=True, contamination=0.005)
model_2.fit(x_train)
predicted_2 = model_2.predict(full_data)
y_true = np.ones(full_data.shape[0]) # create true labels
y_true[-x_test_outliers.shape[0] :] = -1 # last 20 is anomaly
from sklearn.metrics import ConfusionMatrixDisplay
fig = plt.figure(figsize=(10, 6))
ax1 = fig.add_subplot(121)
ax2 = fig.add_subplot(122)
ConfusionMatrixDisplay.from_predictions(y_true, predicted, ax=ax1, colorbar=False)
ConfusionMatrixDisplay.from_predictions(y_true, predicted_2, ax=ax2, colorbar=False)
ax1.set_title("threshold = 0.05")
ax2.set_title("threshold = 0.005")
plt.subplots_adjust(wspace=0.3)
plt.show()
Еще одна идея улучшения качества детекции аномалий — это агрегация результатов разных алгоритмов, т. е. создание ансамбля.
Когда говорят про задачу кластеризации, подразумевают примерно следующее:
У нас есть неразмеченные данные, и мы хотим поделить эти данные по каким-то группам, где в каждой группе будут объекты, похожие друг на друга. Под похожестью как правило понимается выбранная метрика расстояния.
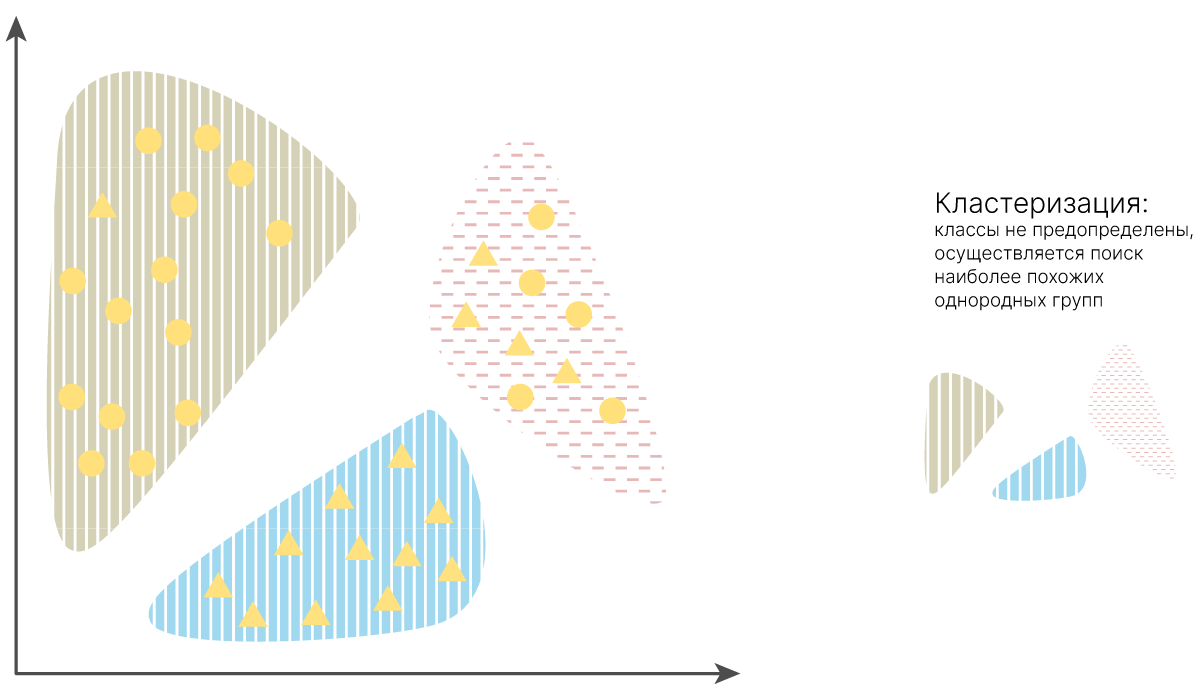
Основные цели кластеризации:
Исследование данных. Мы хотим понять, какие кластеры являются ярко выраженными, и сделать дополнительные выводы о наших данных.
Нахождение аномалий/выбросов. Мы можем искать объекты, которые сильно отличаются от всех остальных и не принадлежат никакому кластеру.
Создание дополнительных признаков. Кластер для объекта уже является дополнительным категориальным признаком.
Непосредственно классификация. Например, можем считать получившиеся кластеры классами и классифицировать объекты таким образом.
Алгоритмов кластеризации существует большое количество, и все работают по-разному, имеют разные параметры для настройки. Мы рассмотрим два наиболее популярных: K-Means 🛠️[doc] и DBSCAN 🛠️[doc].

Особенности K-Means:
import matplotlib.pyplot as plt
import seaborn as sns
from sklearn.cluster import KMeans
from sklearn.datasets import make_blobs, make_moons
x_blobs, _ = make_blobs(n_samples=600, n_features=2, centers=3, random_state=42)
x_moons, _ = make_moons(n_samples=600, noise=0.05, random_state=42)
def plot_kmeans_clustering(ax, data, n_clusters, title):
kmeans = KMeans(n_clusters=n_clusters, n_init=10)
kmeans.fit(data)
kmeans_labels = kmeans.predict(data)
sns.scatterplot(
x=data[:, 0], y=data[:, 1], hue=kmeans_labels, palette="Set2", ax=ax
)
ax.scatter(
kmeans.cluster_centers_[:, 0],
kmeans.cluster_centers_[:, 1],
s=100,
c="red",
marker="o",
)
ax.set_title(title)
ax.set_xlabel("Feature 1")
ax.set_ylabel("Feature 2")
fig, axes = plt.subplots(1, 2, figsize=(12, 6))
plot_kmeans_clustering(
axes[0], x_blobs, n_clusters=3, title="KMeans Clustering - Blobs"
)
plot_kmeans_clustering(
axes[1], x_moons, n_clusters=2, title="KMeans Clustering - Moons"
)
plt.tight_layout()
plt.show()
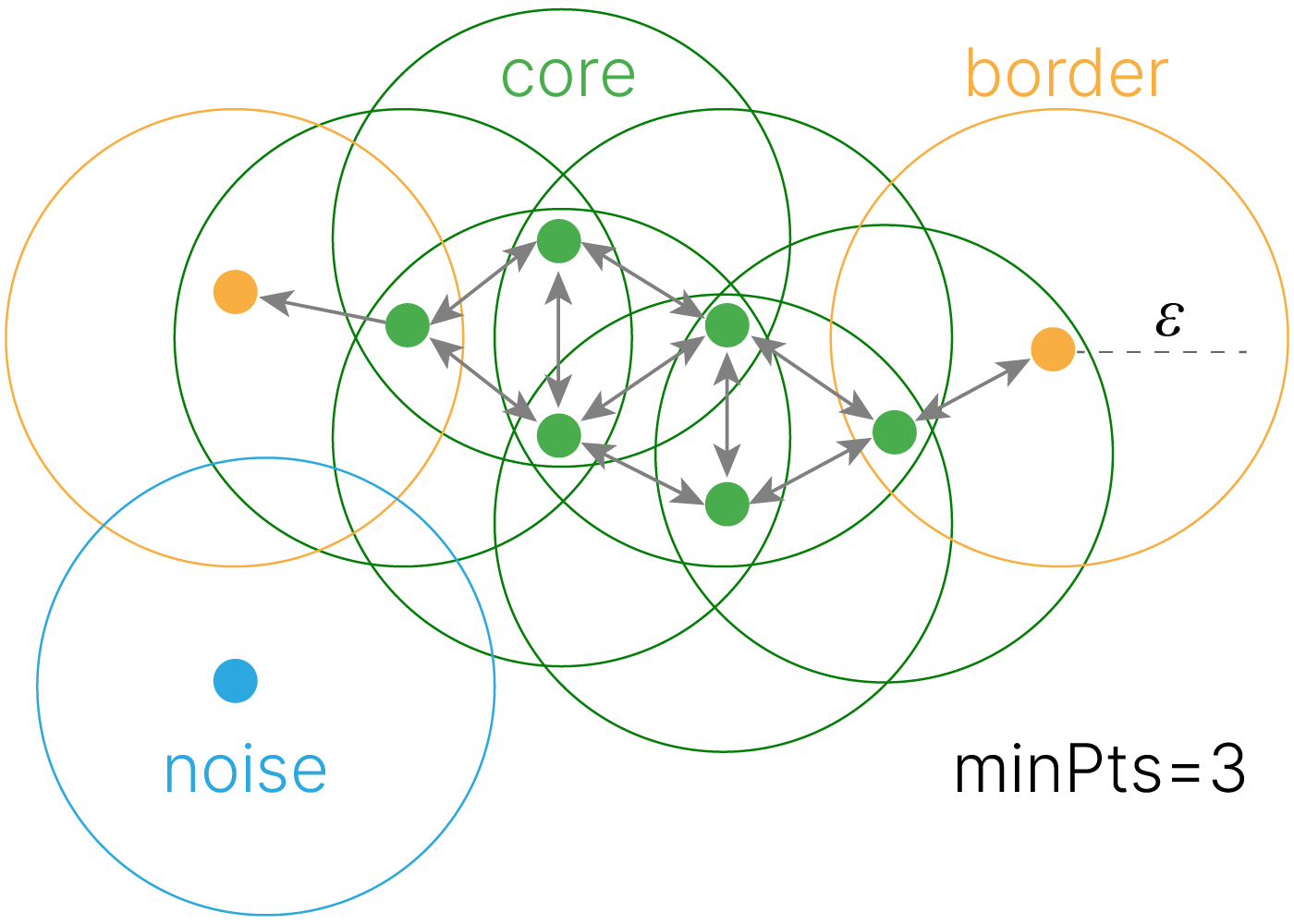
DBSCAN (Density-based spatial clustering of applications with noise, плотностный алгоритм пространственной кластеризации с присутствием шума), как следует из названия, оперирует плотностью данных. На вход требуется матрица близости и два параметра:
eps — радиус $\varepsilon$-окрестности, в которой мы ищем похожие объекты (радиус кружков на картинке).min_samples — количество соседей, которое должен иметь объект, чтобы не быть одиночкой. У нас min_samples=3.Алгоритм выглядит следующим образом:
Иначе:
from sklearn.cluster import DBSCAN
def plot_dbscan_clustering(ax, data, eps, min_samples, title):
dbscan = DBSCAN(eps=eps, min_samples=min_samples)
dbscan_labels = dbscan.fit_predict(data)
sns.scatterplot(
x=data[:, 0], y=data[:, 1], hue=dbscan_labels, palette="Set2", ax=ax
)
ax.set_title(title)
ax.set_xlabel("Feature 1")
ax.set_ylabel("Feature 2")
fig, axes = plt.subplots(1, 2, figsize=(12, 6))
plot_dbscan_clustering(
axes[0], x_blobs, eps=0.5, min_samples=5, title="DBSCAN Clustering - Blobs"
)
plot_dbscan_clustering(
axes[1], x_moons, eps=0.15, min_samples=5, title="DBSCAN Clustering - Moons"
)
plt.tight_layout()
plt.show()
Кодирование признаков — процесс описания данных в численном представлении.
Изначально объекты в нашем датасете могут быть представлены в виде описаний, которые не являются численными или требуют некоторой предобработки. Например:
Большая часть моделей не способна работать с такими представлениями в сыром виде и просто не запустится или будет выдавать неадекватные результаты.
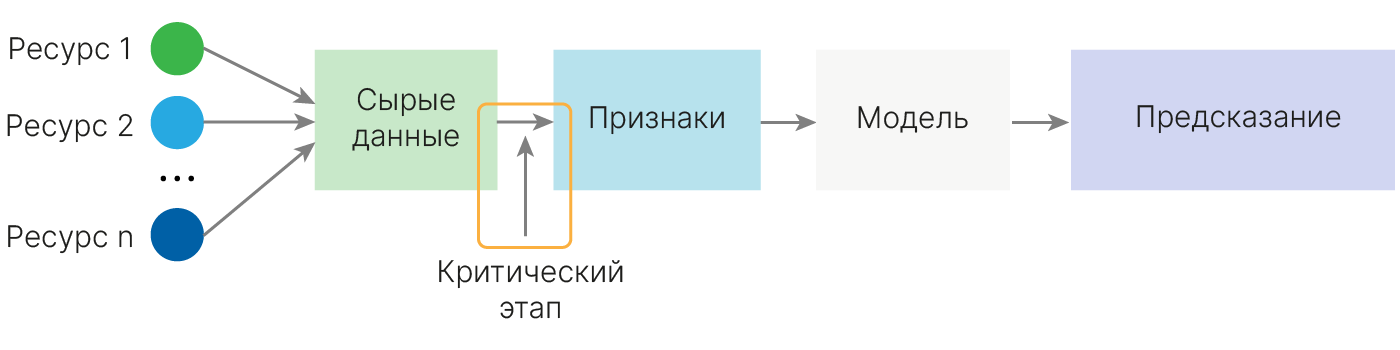
Пример на Titanic
Для иллюстрации будут использованы примеры из книги "Real-World Machine Learning 📚[book]" из открытого репозитория 🐾[git] и датасет Titanic 🛠️[doc].
Данный датасет представляет собой список пассажиров судна. В отличие от датасетов, с которыми мы работали раньше, данные в нем не предобработаны и в сыром виде не могут быть использованы для обучения модели.
import pandas as pd
# Download the data and save it in a variable called data
dataset = pd.read_csv(
"https://edunet.kea.su/repo/EduNet-web_dependencies/datasets/titanic.csv"
) # Load the data using pandas
dataset[:5] # Show the first 5 lines
| PassengerId | Survived | Pclass | Name | Sex | Age | SibSp | Parch | Ticket | Fare | Cabin | Embarked | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 1 | 0 | 3 | Braund, Mr. Owen Harris | male | 22.0 | 1 | 0 | A/5 21171 | 7.2500 | NaN | S |
| 1 | 2 | 1 | 1 | Cumings, Mrs. John Bradley (Florence Briggs Th... | female | 38.0 | 1 | 0 | PC 17599 | 71.2833 | C85 | C |
| 2 | 3 | 1 | 3 | Heikkinen, Miss. Laina | female | 26.0 | 0 | 0 | STON/O2. 3101282 | 7.9250 | NaN | S |
| 3 | 4 | 1 | 1 | Futrelle, Mrs. Jacques Heath (Lily May Peel) | female | 35.0 | 1 | 0 | 113803 | 53.1000 | C123 | S |
| 4 | 5 | 0 | 3 | Allen, Mr. William Henry | male | 35.0 | 0 | 0 | 373450 | 8.0500 | NaN | S |
Если мы попытаемся обучить модель на таких данных, то у нас ничего не выйдет.
from sklearn.ensemble import RandomForestClassifier
x = dataset.drop("Survived", axis=1) # drop target
y = dataset["Survived"] # target
rf = RandomForestClassifier(random_state=42)
try:
rf.fit(x, y)
except ValueError as e:
print(e)
could not convert string to float: 'Braund, Mr. Owen Harris'
Посмотрим на информацию о признаках:
dataset.info()
<class 'pandas.core.frame.DataFrame'> RangeIndex: 891 entries, 0 to 890 Data columns (total 12 columns): # Column Non-Null Count Dtype --- ------ -------------- ----- 0 PassengerId 891 non-null int64 1 Survived 891 non-null int64 2 Pclass 891 non-null int64 3 Name 891 non-null object 4 Sex 891 non-null object 5 Age 714 non-null float64 6 SibSp 891 non-null int64 7 Parch 891 non-null int64 8 Ticket 891 non-null object 9 Fare 891 non-null float64 10 Cabin 204 non-null object 11 Embarked 889 non-null object dtypes: float64(2), int64(5), object(5) memory usage: 83.7+ KB
Традиционно признаки делятся на вещественные и категориальные.
Вещественные признаки бывают:

В датасете Титаник таким параметром будет SibSp — количество братьев/сестер/супругов на борту.
print(dataset["SibSp"].unique())
[1 0 3 4 2 5 8]
Или Parch — количество родителей/детей на борту.
print(dataset["Parch"].unique())
[0 1 2 5 3 4 6]

Разделение условное. Тот же возраст можно посчитать и дискретной переменной (пользователь всегда нам сообщает свои полные года), и непрерывной (возраст можно считать с любой точностью)
Иногда вещественные признаки делят на относительные (считаются относительно чего-то, уже нормированные и т.д.) и интервальные.
dataset[["Age", "Fare"]].head()
| Age | Fare | |
|---|---|---|
| 0 | 22.0 | 7.2500 |
| 1 | 38.0 | 71.2833 |
| 2 | 26.0 | 7.9250 |
| 3 | 35.0 | 53.1000 |
| 4 | 35.0 | 8.0500 |
Попробуем обучить модель на вещественных признаках. PassengerId удалим, т.к. этот признак может приводить к утечке в данных.
from sklearn.model_selection import train_test_split
x = dataset.drop(columns=["Survived", "PassengerId"]) # drop target and id
y = dataset["Survived"] # target
x_train, x_test, y_train, y_test = train_test_split(
x, y, test_size=0.2, random_state=42
)
# drop categorical
x_train_working = x_train.drop(
columns=["Pclass", "Name", "Sex", "Ticket", "Cabin", "Embarked"]
)
x_test_working = x_test.drop(
columns=["Pclass", "Name", "Sex", "Ticket", "Cabin", "Embarked"]
)
rf = RandomForestClassifier(random_state=42)
try:
rf.fit(x_train_working, y_train)
except ValueError as e:
print(e)
x_train_working.head()
Input X contains NaN. RandomForestClassifier does not accept missing values encoded as NaN natively. For supervised learning, you might want to consider sklearn.ensemble.HistGradientBoostingClassifier and Regressor which accept missing values encoded as NaNs natively. Alternatively, it is possible to preprocess the data, for instance by using an imputer transformer in a pipeline or drop samples with missing values. See https://scikit-learn.org/stable/modules/impute.html You can find a list of all estimators that handle NaN values at the following page: https://scikit-learn.org/stable/modules/impute.html#estimators-that-handle-nan-values
| Age | SibSp | Parch | Fare | |
|---|---|---|---|---|
| 331 | 45.5 | 0 | 0 | 28.5000 |
| 733 | 23.0 | 0 | 0 | 13.0000 |
| 382 | 32.0 | 0 | 0 | 7.9250 |
| 704 | 26.0 | 1 | 0 | 7.8542 |
| 813 | 6.0 | 4 | 2 | 31.2750 |
Проблема в том, что Age указан только для 714 пассажиров из 891. Его мы пока тоже удалим.
from sklearn.metrics import accuracy_score
x_train_working = x_train_working.drop(columns=["Age"])
x_test_working = x_test_working.drop(columns=["Age"])
rf = RandomForestClassifier(random_state=42)
rf.fit(x_train_working, y_train)
y_pred = rf.predict(x_test_working)
print(accuracy_score(y_test, y_pred))
0.6759776536312849
Точность предсказания 67.6%. Для улучшения качества попробуем добавить другие признаки.
Значение — принадлежность к какой-то из категорий. Традиционно делятся на сильно отличающиеся по свойствам:

В датасете Титаник таким признаком будет класс. Мы можем сказать, что первый класс лучше третьего, но не можем сказать, что сумма первого и второго даст третий.
print(dataset["Pclass"].unique())
[3 1 2]

Важно понимать, что могут быть ситуации, когда один и тот же признак может быть упорядоченным или неупорядоченным, в зависимости от задачи и наших знаний о ней. Например, цвет в спектре определяется длиной волны, и в физической задаче цвета можно упорядочить.
В датасете Титаник таким признаком является Embarked (порт посадки).
print(dataset["Embarked"].unique())
# C = Cherbourg; Q = Queenstown; S = Southampton
['S' 'C' 'Q' nan]
Часто мы сталкиваемся с бинарными категориальными признаками, для которых известны только две возможные категории (например, биологический пол человека).
print(dataset["Sex"].unique())
['male' 'female']
Существует много подходов к кодированию признакового пространства. Каждый подход имеет свои особенности. Способ кодирования зависит от используемой модели.
В Label encoding каждой категории признака однозначно сопоставляется число. Данный подход хорошо работает для упорядоченных (ординальных) признаков.
Если признак неупорядоченный (номинальный), то могут возникнуть проблемы. Например, если мы обозначим: $$Уж = 1$$ $$Ёж = 2$$ $$Белка = 3$$ То в некоторых моделях (линейных), может возникнуть ложная зависимость:
$$Уж+Ёж = Белка$$что не является свойством данных.
Кроме того, мы не можем сказать, что уж “больше” ежа и сравнить его с белкой, но обучаемая модель про это не знает и будет пытаться их сравнить. Это может привести к низкому качеству модели и выучиванию неправильной информации.
Например, дерево решений для выделения одной категории должно будет проделать несколько сравнений.
Некоторые модели (например, LightGBM 🛠️[doc]) могут автоматически подбирать кодировку для категориальных признаков, если предоставить им информацию о том, что признак категориальный, для других моделей это нужно делать вручную.
У нас есть упорядоченный категориальный признак — класс, которым ехал пассажир. Добавим его к данным.
x_train_working["Pclass"] = x_train["Pclass"]
x_test_working["Pclass"] = x_test["Pclass"]
x_train_working[:5]
| SibSp | Parch | Fare | Pclass | |
|---|---|---|---|---|
| 331 | 0 | 0 | 28.5000 | 1 |
| 733 | 0 | 0 | 13.0000 | 2 |
| 382 | 0 | 0 | 7.9250 | 3 |
| 704 | 1 | 0 | 7.8542 | 3 |
| 813 | 4 | 2 | 31.2750 | 3 |
На практике часто используется one-hot encoding. Вместо одного категориального признака $X$ создается набор бинарных категориальных признаков, которые отвечают на вопрос $X == C$, где $C$ перебирает все возможные значения категориального признака.
Теперь, чтобы выбрать конкретное значение категориального признака, дереву решений достаточно задать один вопрос.
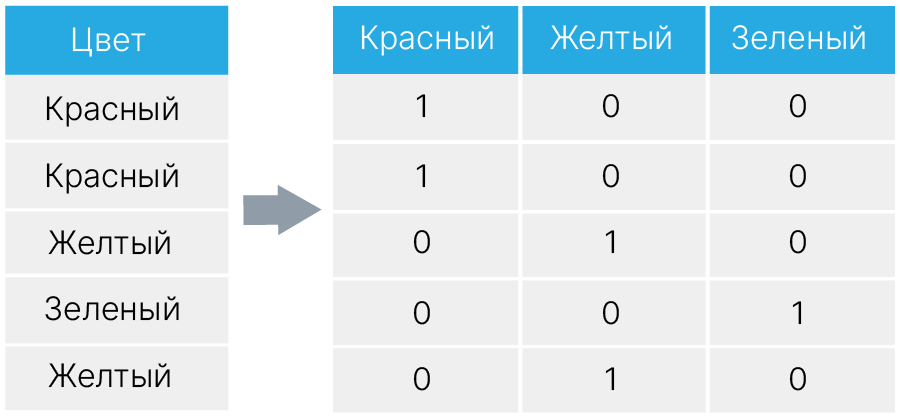
У такой схемы есть ряд недостатков:
Подробнее:
[blog] ✏️ Think twice before dropping that first one-hot encoded column.
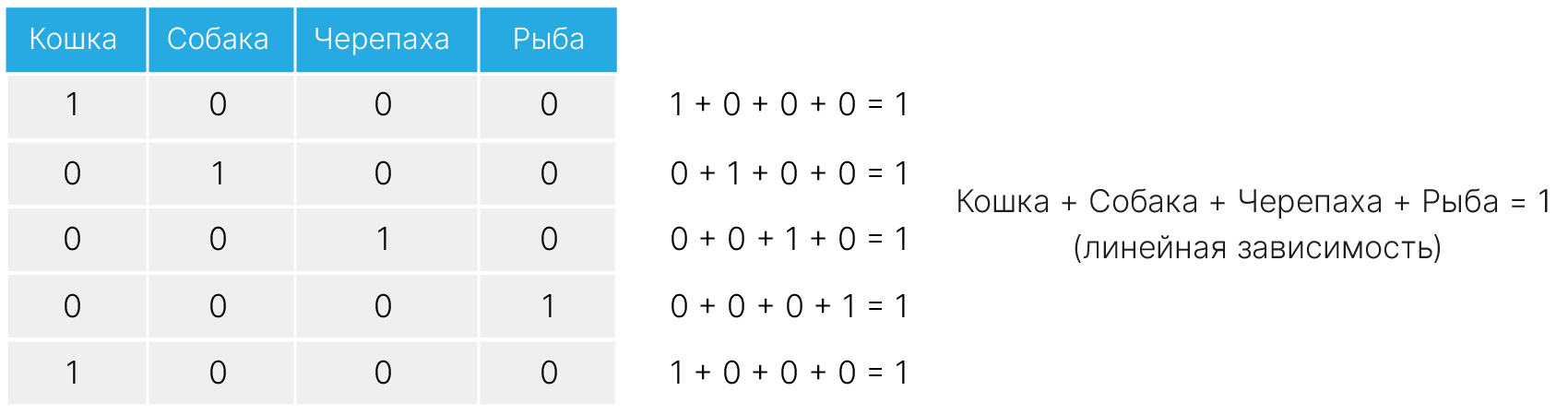
Поэтому одну из категорий могут исключить при кодировании. Например, в примере выше можно исключить Рыбу, ведь если все три других признака-категории равны 0, то точно верно, что категория — Рыба.
У нас есть два признака с ограниченным количеством значений: Sex и Embarked.
Пол закодируем male = 1 female = 0.
x_train_working["Sex"] = x_train["Sex"].map({"male": 1, "female": 0})
x_test_working["Sex"] = x_test["Sex"].map({"male": 1, "female": 0})
x_train_working[:5]
| SibSp | Parch | Fare | Pclass | Sex | |
|---|---|---|---|---|---|
| 331 | 0 | 0 | 28.5000 | 1 | 1 |
| 733 | 0 | 0 | 13.0000 | 2 | 1 |
| 382 | 0 | 0 | 7.9250 | 3 | 1 |
| 704 | 1 | 0 | 7.8542 | 3 | 1 |
| 813 | 4 | 2 | 31.2750 | 3 | 0 |
Еще один способ, который показал свою эффективность — это кодирование категориальной переменной по количеству встречаемости в данных.
| category | new_feature |
|---|---|
| food | 3 |
| equipment | 2 |
| food | 3 |
| food | 3 |
| equipment | 2 |
| clothes | 1 |
Его просто и быстро реализовать. Здесь также присутствует логика порядка, который возникает естественным образом. Чем больше число, тем чаще встречается категория. Также здесь отсутствует ложная линейная зависимость: в нашем случае equipment + clothes = food, и для нас это действительно так, потому что наше кодирование отражает связь между категориями через их частоту.
Дополнительно стоит добавить шум к данным, чтобы избежать ситуации, когда разные категории кодируются одинаковым числом.
| category | new_feature |
|---|---|
| food | 2.3 |
| equipment | 2.04 |
| food | 2.3 |
| clothes | 2.6 |
| equipment | 2.04 |
| clothes | 2.6 |
Можем кодировать категориальный признак по какой-то статистике вещественного. Например, при прогнозировании покупок в интернет магазине может оказаться разумным кодировать категории товаров их средней ценой.
| category | product | price | new_feature |
|---|---|---|---|
| food | pizza | 30 | 17 |
| equipment | hammer | 140 | 170 |
| food | cucumber | 4 | 17 |
| clothes | boots | 100 | 60 |
| equipment | helmet | 200 | 170 |
| clothes | gloves | 20 | 60 |
В Target encoding каждая категория кодируется численным параметром, характеризующим то, что мы предсказываем. Например, можно каждую категорию категориального признака заменять на среднее целевого значения (mean target).
При этом может возникнуть проблема переобучения: для редких классов модель может научиться копировать значение mean target категориального признака в ответ, игнорируя другие признаки.
Как с этим бороться:
[git] 🐾 Курс "Прикладные задачи анализа данных", Генерация признаков.
Так как у нас в качестве модели используется случайный лес, для Embarked будем использовать Label encoding. Мы не будем считать среднее. Используем Target, чтобы упорядочить метки. Посмотрим, какой процент выживших для каждого порта. Смотреть будем только на train выборке. Информация из test выборки не должна попасть в модель!
import seaborn as sns
from matplotlib import pyplot as plt
plt.figure(figsize=(8, 4))
train_df = x_train.copy()
train_df["Survived"] = y_train
sns.barplot(x="Embarked", y="Survived", data=train_df)
plt.show()
Получили количество выживших $S<Q<C$. Упорядочим метки соответственно.
import numpy as np
emb = {np.nan: 0, "S": 0, "Q": 1, "C": 2}
x_train_working["Embarked"] = x_train["Embarked"].map(emb)
x_test_working["Embarked"] = x_test["Embarked"].map(emb)
x_train_working[5:10]
| SibSp | Parch | Fare | Pclass | Sex | Embarked | |
|---|---|---|---|---|---|---|
| 118 | 0 | 1 | 247.5208 | 1 | 1 | 2 |
| 536 | 0 | 0 | 26.5500 | 1 | 1 | 0 |
| 361 | 1 | 0 | 27.7208 | 2 | 1 | 2 |
| 29 | 0 | 0 | 7.8958 | 3 | 1 | 0 |
| 55 | 0 | 0 | 35.5000 | 1 | 1 | 0 |
У нас осталось еще 4 признака, с которыми непонятно, что делать: Name, Ticket, Cabin, Age.
Рассмотрим поле Name. Это поле может нести информацию о поле, социальном статусе, происхождении, национальности, возрасте и т.д. Можно построить модели, которые будут это оценивать и отражать. Мы будем работать с числовыми представлениями текста в нашем курсе. Но пока мы можем использовать метод "пристального вглядывания" в данные.
x_train.Name[:5]
331 Partner, Mr. Austen 733 Berriman, Mr. William John 382 Tikkanen, Mr. Juho 704 Hansen, Mr. Henrik Juul 813 Andersson, Miss. Ebba Iris Alfrida Name: Name, dtype: object
В именах есть информация о социальном статусе:
и т.д.
Первые 4 встречаются чаще.
Извлечем все возможные титулы, они начинаются с пробела и заканчиваются точкой.
titles = dataset.Name.str.extract(" ([A-Za-z]+)\.", expand=False).unique()
print(titles)
['Mr' 'Mrs' 'Miss' 'Master' 'Don' 'Rev' 'Dr' 'Mme' 'Ms' 'Major' 'Lady' 'Sir' 'Mlle' 'Col' 'Capt' 'Countess' 'Jonkheer']
Сгруппируем некоторые с похожими значениями. Редкие запишем в нулевой класс.
title_mapping = {"Mr": 4, "Miss": 3, "Mrs": 2, "Master": 1}
# get title from name and encode by title_mapping, another titles fill zero
x_train_working["Title"] = (
x_train.Name.str.extract(" ([A-Za-z]+)\.", expand=False)
.map(title_mapping)
.fillna(0)
)
x_test_working["Title"] = (
x_test.Name.str.extract(" ([A-Za-z]+)\.", expand=False).map(title_mapping).fillna(0)
)
x_train_working[:5]
| SibSp | Parch | Fare | Pclass | Sex | Embarked | Title | |
|---|---|---|---|---|---|---|---|
| 331 | 0 | 0 | 28.5000 | 1 | 1 | 0 | 4.0 |
| 733 | 0 | 0 | 13.0000 | 2 | 1 | 0 | 4.0 |
| 382 | 0 | 0 | 7.9250 | 3 | 1 | 0 | 4.0 |
| 704 | 1 | 0 | 7.8542 | 3 | 1 | 0 | 4.0 |
| 813 | 4 | 2 | 31.2750 | 3 | 0 | 0 | 3.0 |
При работе с датой и временем мы можем представить дату и время в виде числа. Один из способов такого представления Unix Timestamp 🛠️[doc] (количество секунд, прошедших с 1 января 1970-го года). Для ряда задач важна цикличность времени. Например, загруженность линии метро будет зависеть от времени дня (цикл 24 часа), дня недели (цикл 7 дней) и нерабочих праздничных дней (цикл год). Для прогнозирования количества электроэнергии, выработанной солнечной батареей, важно будет время дня (цикл 24 часа) и время года (цикл год).
В случае с такими признаками, как день недели или время суток, мы сталкиваемся с проблемой того, что нам нужно предложить кодирование, которое будет учитывать, что понедельник близок к воскресенью так же, как и ко вторнику, и т.д.
Давайте нанесем наши категории, например, дни недели, на окружность. Как это сделать? Пусть понедельнику соответствует 1, а воскресенью — 7. Далее посчитаем два таких вспомогательных признака по следующим формулам
weekdays = np.arange(1, 8) # create an array of weekdays
print(weekdays)
sina = np.sin(weekdays * np.pi * 2 / np.max(weekdays)) # feature 1
cosa = np.cos(weekdays * np.pi * 2 / np.max(weekdays)) # feature 2
[1 2 3 4 5 6 7]
plt.figure(figsize=(6, 6)) # Decide figure size
plt.scatter(sina, cosa) # Plot scatter of feature 1 vs feature 2
for i, z in enumerate(
("Mon", "Tue", "Wed", "Thu", "Fri", "Sat", "Sun")
): # for each day in a week
plt.text(sina[i], cosa[i], s=z) # add text labels to plot
Теперь расстояния между понедельником и вторником и воскресеньем и понедельником одинаковые:
dist_mon_tue = (sina[1] - sina[0]) ** 2 + (
cosa[1] - cosa[0]
) ** 2 # distance between Monday and Tuesday
dist_sun_mon = (sina[6] - sina[0]) ** 2 + (
cosa[6] - cosa[0]
) ** 2 # distance between Sunday and Monday
print("Distance between Mon-Tue = %.2f" % dist_mon_tue)
print("Distance between Sun-Mon = %.2f" % dist_sun_mon)
Distance between Mon-Tue = 0.75 Distance between Sun-Mon = 0.75
То же самое верно и для любых отстоящих друг от друга на одинаковое число дней
dist_mon_wed = (sina[2] - sina[0]) ** 2 + (
cosa[2] - cosa[0]
) ** 2 # distance between Monday and Wednesday
dist_fri_sun = (sina[4] - sina[6]) ** 2 + (
cosa[4] - cosa[6]
) ** 2 # distance between Friday and Sunday
print("Distance between Mon-Wed = %.2f" % dist_mon_wed)
print("Distance between Fri-Sun = %.2f" % dist_fri_sun)
Distance between Mon-Wed = 2.45 Distance between Fri-Sun = 2.45
Таким образом, циклические признаки можно кодировать парой признаков (sin и cos), полученных по схеме, описанной выше.
Проблемы подхода:
Такое кодирование может создать сложности для деревьев решений, т.к. они работают с одним признаком за раз.
Надо понимать, что важность исходной категориальной фичи неочевидным образом делится между двумя полученными из нее таким образом фичами.
В некоторых задачах one-hot работает лучше.
Для ряда задач может быть неважно конкретное значение признака. Важнее может оказаться факт превышения порога или наличия значения.
Например, уровень сахара крови выше $11.1$ ммоль/л может говорить о наличии у пациента сахарного диабета, что повлияет на результат лечения. А наличие высшего образования больше влияет на платежеспособность, чем средний балл диплома.
Для таких признаков можно попробовать использовать бинаризацию: превращение вещественного признака в бинарный по принципу “есть ли значение” или “больше ли значение определенного порога”.
Бинаризируем уровень сахара в крови.
from sklearn.preprocessing import Binarizer
# fmt: off
x = np.array([[1, 12],
[2, 7.6],
[3, 8.4],
[4, 13.5],
[5, 6.3]])
# fmt: on
transformer = Binarizer(threshold=11.1)
binarized = transformer.transform(np.expand_dims(x[:, 1], axis=1))
x_binarized = np.concatenate((x, binarized), axis=1)
print(x_binarized)
[[ 1. 12. 1. ] [ 2. 7.6 0. ] [ 3. 8.4 0. ] [ 4. 13.5 1. ] [ 5. 6.3 0. ]]
Нам могут быть не интересны точные значения (например, что видео набрало 1000 лайков, а не 1001).
К тому же, число просмотров/лайков некоторых видео может быть очень большим в сравнении с остальными. В итоге часть значений у нас встречается часто, а часть — очень редко, что негативно скажется на результате работы модели.
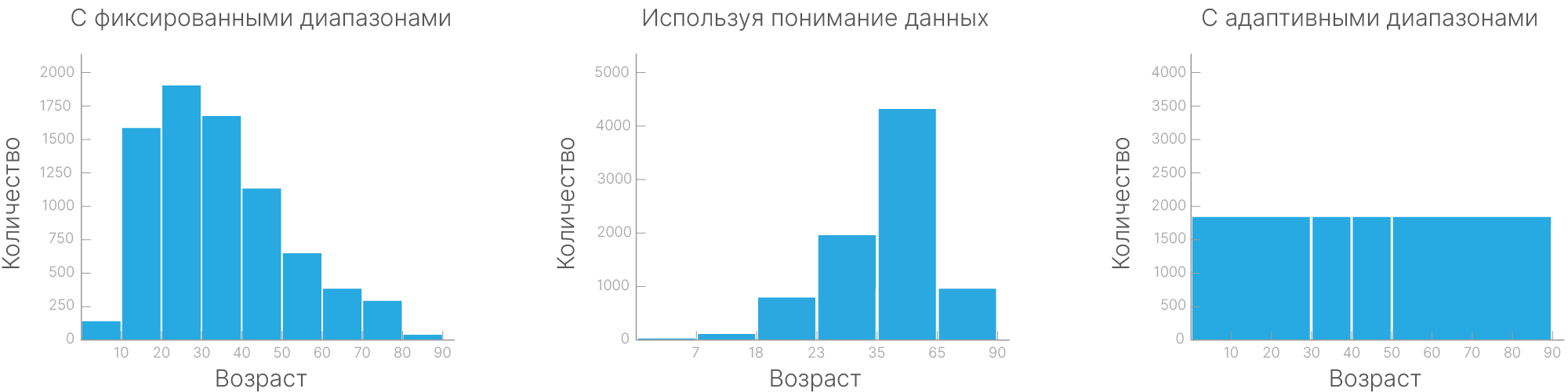
Бинирование — это метод группировки вещественных признаков в несколько категорий, определяемых диапазонами значений. При этом категория может кодироваться средним или медианным значением признака в диапазоне данной категории.
Способы деления данных:
Фиксированными диапазонами. Делим наши значения по диапазонам фиксированной длины. Так часто поступают с возрастом.
Используя понимание данных. Длина диапазона не всегда обязана быть кратна определенному ровному значению. В социальных исследованиях может быть полезным разделение на возрастные группы, которые определяются занятостью: школьники, студенты, выпускники, пенсионеры и т.д.
Адаптивными диапазонами. Бинирование с фиксированной длиной бина не поможет справиться с редкими значениями. В этой ситуации помогает бинирование, например, по квартилям — когда границы бина расставляются таким образом, чтобы между ними помещалась $1/4$ выборки.
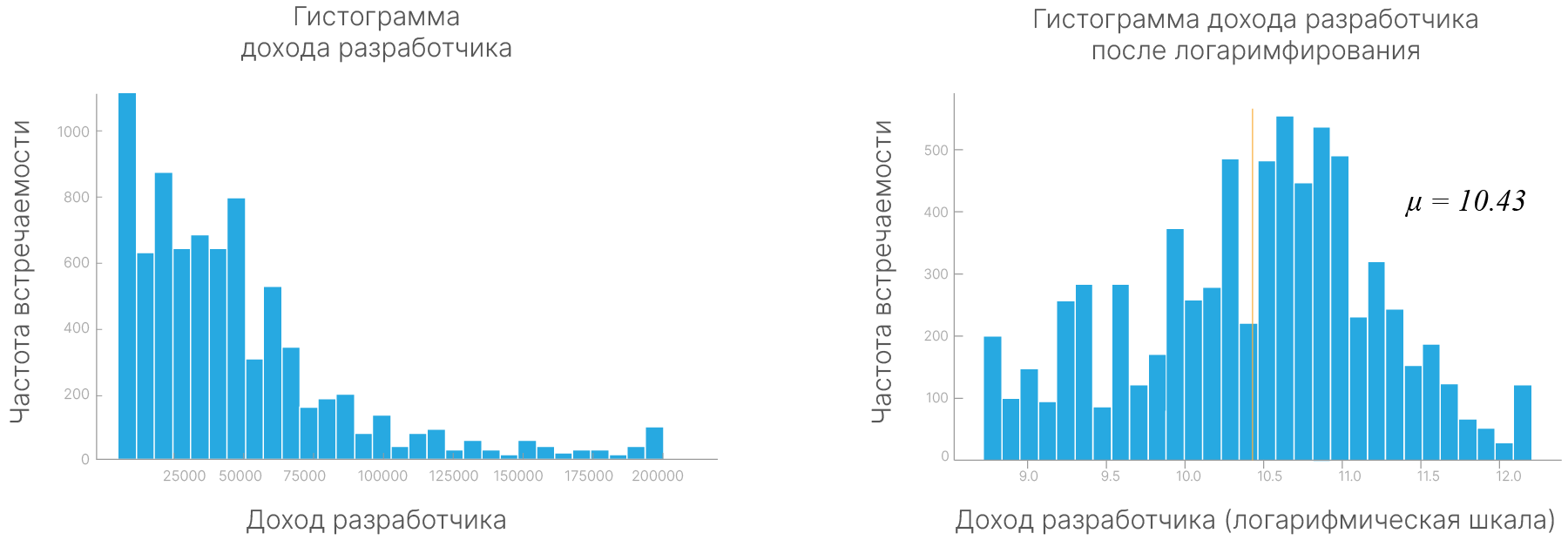
Логарифмическое преобразование — это применение логарифма к значениям признака. Это может быть полезным, если данные имеют сильно смещенное распределение, например, экспоненциальное. Логарифмическое преобразование может сделать распределение более близким к нормальному.
Квадратный корень. Взятие квадратного корня из значений признаков может помочь справиться с сильно смещенными данными. Он уменьшает влияние больших значений и увеличивает влияние меньших значений.
Преобразование Бокса-Кокса (Box-Cox Transform ✏️[blog]) — это семейство степенных преобразований, которое может адаптироваться к различным распределениям данных, общей целью которых является придать данным вид, более похожий на нормальное распределение, с которым работает бoльшее число моделей и сходимость которого лучше.
from scipy import stats
np.random.seed(42)
data = np.random.exponential(scale=2, size=1000)
plt.figure(figsize=(12, 4))
gs = plt.GridSpec(2, 3, height_ratios=[2, 1])
plt.subplot(gs[0, 0])
sns.scatterplot(x=np.arange(len(data)), y=data, s=10)
plt.title("Original Data")
plt.subplot(gs[1, 0])
sns.histplot(data, bins=30)
data_log = np.log(data)
plt.subplot(gs[0, 1])
sns.scatterplot(x=np.arange(len(data_log)), y=data_log, s=10)
plt.title("Log Transformation")
plt.subplot(gs[1, 1])
sns.histplot(data_log, bins=30)
data_boxcox, _ = stats.boxcox(data)
plt.subplot(gs[0, 2])
sns.scatterplot(x=np.arange(len(data_boxcox)), y=data_boxcox, s=10)
plt.title("Box-Cox Transformation")
plt.subplot(gs[1, 2])
sns.histplot(data_boxcox, bins=30)
plt.tight_layout()
plt.show()
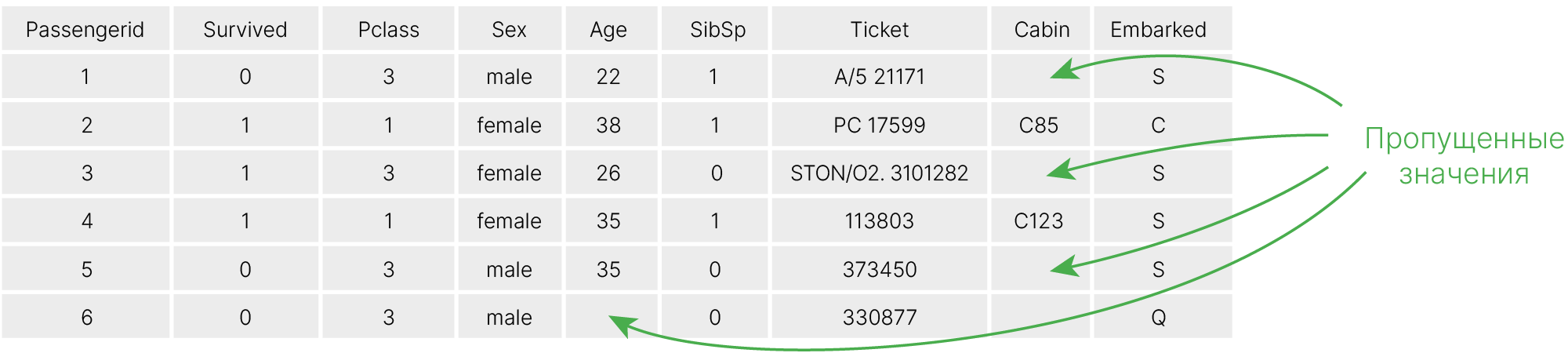
Со всеми признаками производятся математические операции, поэтому для большинства моделей (деревья решений могут работать с пропущенными значениями, Meaning of Surrogate Split ✏️[blog]) отсутствие значений просто не дает производить эти операции.
Есть два основных подхода для работы с пропущенными значениями:
Удалить все объекты с пропущенными значениями.
На место пропущенных значений записать какие-то числа (заполнить пропущенные значения)
Иногда мы можем просто удалить объекты с пропущенными значениями, но если у нас мало данных и получение большего количества данных дорого/долго/невозможно, то нам нужно заполнить пропуски какими-то значениями.
Существует много способов заполнения пропусков значениями.
Рассмотрим наиболее популярные:
Заполнить константой. Можно предположить, что данные несложные, а признаки не зависят друг от друга, и заполнить пропущенные значения средними значениями соответствующего признака. Например, если средний возраст людей 30 лет, то для людей, чей возраст неизвестен, будем писать это же значение. Но в некоторых ситуациях такое заполнение может только навредить.
Предсказывать признаки моделью. Используем признак с пропущенными значениями как целевую переменную и на основе других заполненных признаков предсказываем пропущенные значения. Можем использовать алгоритм, который умеет справляться с пропусками. Один из таких алгоритмов — k-NN. Можно брать ближайших соседей по известным признакам и на место неизвестного признака подставлять среднее значение соседей.
Самостоятельно придумывать логику заполнения. Если мы имеем экспертное знание о природе пропусков, можем сами написать логику заполнения какими-то значениями.
В датасете Titanic отстался важный признак Age, который имеет пропуски. Посмотрим, как он связан с выживаемостью.
train_df = x_train.copy()
train_df["Survived"] = y_train
plt.figure(figsize=(10, 6))
age_survival_plot = sns.scatterplot(
data=train_df.groupby(["Age"], as_index=False)["Survived"].agg("sum"),
x="Age",
y="Survived",
hue="Survived",
s=70,
palette={
0: "#27a9e1",
1: "#ffab40",
2: "#ffab40",
3: "#ffab40",
4: "#ffab40",
5: "#ffab40",
6: "#49AD4D",
7: "#49AD4D",
8: "#49AD4D",
9: "#49AD4D",
11: "#49AD4D",
15: "#49AD4D",
},
)
age_survival_plot.set_title("Survived by Age")
age_survival_plot.set_xlabel("Age")
age_survival_plot.set_ylabel("Num of passengers")
custom_legend = [
plt.Line2D([0], [0], marker="o", color="w", markerfacecolor=color, markersize=10)
for color in ["#27a9e1", "#ffab40", "#49AD4D"]
]
age_survival_plot.legend(
custom_legend, ["0", "1-5", "6+"], title="Survived", loc="upper right"
)
plt.show()
Видим, что связь есть. Обучим модель без этого признака и посмотрим на результат:
x_train_working.head(3)
| SibSp | Parch | Fare | Pclass | Sex | Embarked | Title | |
|---|---|---|---|---|---|---|---|
| 331 | 0 | 0 | 28.500 | 1 | 1 | 0 | 4.0 |
| 733 | 0 | 0 | 13.000 | 2 | 1 | 0 | 4.0 |
| 382 | 0 | 0 | 7.925 | 3 | 1 | 0 | 4.0 |
results = dict()
rf = RandomForestClassifier(random_state=42)
rf.fit(x_train_working, y_train)
y_pred = rf.predict(x_test_working)
results["no feature"] = accuracy_score(y_test, y_pred).round(3)
print(f"Accuracy without feature 'Age': {results['no feature']}")
Accuracy without feature 'Age': 0.816
Заполним пропуски своей эвристикой. Вспомним, что в поле Title зашита общая информация о возрасте, посчитаем матожидание и дисперсию возраста для Title и сгенерируем недостающие значения.
np.random.seed(42)
train_df = x_train.copy()
train_df["Title"] = x_train_working["Title"]
mean = {}
std = {}
for title in range(5):
data = train_df.loc[train_df["Title"] == title]
mean[title] = data["Age"].mean()
std[title] = data["Age"].std()
def add_age_val(data, mean, std):
for i, row in data.iterrows():
if np.isnan(row["Age"]):
title = int(row["Title"])
data.loc[i, "Age"] = round(
np.random.uniform(
low=int(mean[title] - std[title]),
high=int(mean[title] + std[title]),
),
1,
)
return data
x_train_working["Age"] = x_train["Age"]
x_test_working["Age"] = x_test["Age"]
x_train_working = add_age_val(x_train_working, mean, std)
x_test_working = add_age_val(x_test_working, mean, std)
x_train_working[:5]
| SibSp | Parch | Fare | Pclass | Sex | Embarked | Title | Age | |
|---|---|---|---|---|---|---|---|---|
| 331 | 0 | 0 | 28.5000 | 1 | 1 | 0 | 4.0 | 45.5 |
| 733 | 0 | 0 | 13.0000 | 2 | 1 | 0 | 4.0 | 23.0 |
| 382 | 0 | 0 | 7.9250 | 3 | 1 | 0 | 4.0 | 32.0 |
| 704 | 1 | 0 | 7.8542 | 3 | 1 | 0 | 4.0 | 26.0 |
| 813 | 4 | 2 | 31.2750 | 3 | 0 | 0 | 3.0 | 6.0 |
Посмотрим, что получилось на обработанных данных.
rf = RandomForestClassifier(random_state=42)
rf.fit(x_train_working, y_train)
y_pred = rf.predict(x_test_working)
results["by title"] = accuracy_score(y_test, y_pred).round(3)
print(f"Accuracy with hand-filled feature 'Age': {results['by title']}")
Accuracy with hand-filled feature 'Age': 0.821
x_train_working["Age"] = x_train["Age"]
x_test_working["Age"] = x_test["Age"]
from sklearn.experimental import enable_iterative_imputer
from sklearn.impute import IterativeImputer
imputer = IterativeImputer()
imputer.fit(x_train_working)
x_train_working = imputer.transform(x_train_working)
x_test_working = imputer.transform(x_test_working)
rf = RandomForestClassifier(random_state=42)
rf.fit(x_train_working, y_train)
y_pred = rf.predict(x_test_working)
results["imputer"] = accuracy_score(y_test, y_pred).round(3)
print(f"Accuracy with feature 'Age' filled with IterativeImputer: {results['imputer']}")
Accuracy with feature 'Age' filled with IterativeImputer: 0.832
Сравним результаты при разном заполнении пропусков
pd.DataFrame([results], index=["accuracy"])
| no feature | by title | imputer | |
|---|---|---|---|
| accuracy | 0.816 | 0.821 | 0.832 |
В нашем случае IterativeImputer 🛠️[doc] дал наилучший результат. Хорошая практика — использовать данный метод как быстрый бейзлайн по заполнению пропусков.
Важно помнить, что пропуски в данных не всегда являются отсутствием значений, иногда пропуски могут быть обозначены совершенно разными способами: 0, -, (пусто), 'отсутствует', None, nan, и другие. Поэтому нужно приводить пропуски к единому формату, с которым удобно работать в дальнейшем, например np.nan.
Разведочный анализ данных (exploratory data analysis, EDA) — это процесс изучения данных с целью понимания их свойств. Его используют для поиска закономерностей, нахождения аномалий, создания новых признаков.
Это творческий процесс, и не существует единого рецепта, как это делать. Более того, разбору одной задачи можно посвятить лекцию. Наша задача в этом блоке — познакомиться с основными инструментами для анализа.
Будем работать с датасетом Wine Quality 🛠️[doc] по определению цвета вина в зависимости от его характеристик.
Загрузим датасет и сразу посмотрим, как он выглядит:
import pandas as pd
dataset = pd.read_csv(
"https://edunet.kea.su/repo/EduNet-web_dependencies/datasets/wine_quality_data.csv"
)
dataset
| fixed_acidity | volatile_acidity | citric_acid | residual_sugar | chlorides | free_sulfur_dioxide | total_sulfur_dioxide | density | pH | sulphates | alcohol | quality | is_red | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 7.4 | 0.70 | 0.00 | 1.9 | 0.076 | 11.0 | 34.0 | 0.99780 | 3.51 | 0.56 | 9.4 | 5 | 0 |
| 1 | 7.8 | 0.88 | 0.00 | 2.6 | 0.098 | 25.0 | 67.0 | 0.99680 | 3.20 | 0.68 | 9.8 | 5 | 0 |
| 2 | 7.8 | 0.76 | 0.04 | 2.3 | 0.092 | 15.0 | 54.0 | 0.99700 | 3.26 | 0.65 | 9.8 | 5 | 0 |
| 3 | 11.2 | 0.28 | 0.56 | 1.9 | 0.075 | 17.0 | 60.0 | 0.99800 | 3.16 | 0.58 | 9.8 | 6 | 0 |
| 4 | 7.4 | 0.70 | 0.00 | 1.9 | 0.076 | 11.0 | 34.0 | 0.99780 | 3.51 | 0.56 | 9.4 | 5 | 0 |
| ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... |
| 6492 | 6.2 | 0.21 | 0.29 | 1.6 | 0.039 | 24.0 | 92.0 | 0.99114 | 3.27 | 0.50 | 11.2 | 6 | 1 |
| 6493 | 6.6 | 0.32 | 0.36 | 8.0 | 0.047 | 57.0 | 168.0 | 0.99490 | 3.15 | 0.46 | 9.6 | 5 | 1 |
| 6494 | 6.5 | 0.24 | 0.19 | 1.2 | 0.041 | 30.0 | 111.0 | 0.99254 | 2.99 | 0.46 | 9.4 | 6 | 1 |
| 6495 | 5.5 | 0.29 | 0.30 | 1.1 | 0.022 | 20.0 | 110.0 | 0.98869 | 3.34 | 0.38 | 12.8 | 7 | 1 |
| 6496 | 6.0 | 0.21 | 0.38 | 0.8 | 0.020 | 22.0 | 98.0 | 0.98941 | 3.26 | 0.32 | 11.8 | 6 | 1 |
6497 rows × 13 columns
Можем сделать первые выводы об объеме датасета и количестве признаков.
Теперь посмотрим более детально, используя встроенные методы pandas:
dataset.info()
<class 'pandas.core.frame.DataFrame'> RangeIndex: 6497 entries, 0 to 6496 Data columns (total 13 columns): # Column Non-Null Count Dtype --- ------ -------------- ----- 0 fixed_acidity 6497 non-null float64 1 volatile_acidity 6497 non-null float64 2 citric_acid 6497 non-null float64 3 residual_sugar 6497 non-null float64 4 chlorides 6497 non-null float64 5 free_sulfur_dioxide 6497 non-null float64 6 total_sulfur_dioxide 6497 non-null float64 7 density 6497 non-null float64 8 pH 6497 non-null float64 9 sulphates 6497 non-null float64 10 alcohol 6497 non-null float64 11 quality 6497 non-null int64 12 is_red 6497 non-null int64 dtypes: float64(11), int64(2) memory usage: 660.0 KB
Видим, что нет пропусков. Смотрим, все ли признаки соответствуют своему Dtype, также можем оценить размер используемой памяти.
Посмотрим описательные статистики по признакам:
dataset.describe()
| fixed_acidity | volatile_acidity | citric_acid | residual_sugar | chlorides | free_sulfur_dioxide | total_sulfur_dioxide | density | pH | sulphates | alcohol | quality | is_red | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| count | 6497.000000 | 6497.000000 | 6497.000000 | 6497.000000 | 6497.000000 | 6497.000000 | 6497.000000 | 6497.000000 | 6497.000000 | 6497.000000 | 6497.000000 | 6497.000000 | 6497.000000 |
| mean | 7.215307 | 0.339666 | 0.318633 | 5.443235 | 0.056034 | 30.525319 | 115.744574 | 0.994697 | 3.218501 | 0.531268 | 10.491801 | 5.818378 | 0.753886 |
| std | 1.296434 | 0.164636 | 0.145318 | 4.757804 | 0.035034 | 17.749400 | 56.521855 | 0.002999 | 0.160787 | 0.148806 | 1.192712 | 0.873255 | 0.430779 |
| min | 3.800000 | 0.080000 | 0.000000 | 0.600000 | 0.009000 | 1.000000 | 6.000000 | 0.987110 | 2.720000 | 0.220000 | 8.000000 | 3.000000 | 0.000000 |
| 25% | 6.400000 | 0.230000 | 0.250000 | 1.800000 | 0.038000 | 17.000000 | 77.000000 | 0.992340 | 3.110000 | 0.430000 | 9.500000 | 5.000000 | 1.000000 |
| 50% | 7.000000 | 0.290000 | 0.310000 | 3.000000 | 0.047000 | 29.000000 | 118.000000 | 0.994890 | 3.210000 | 0.510000 | 10.300000 | 6.000000 | 1.000000 |
| 75% | 7.700000 | 0.400000 | 0.390000 | 8.100000 | 0.065000 | 41.000000 | 156.000000 | 0.996990 | 3.320000 | 0.600000 | 11.300000 | 6.000000 | 1.000000 |
| max | 15.900000 | 1.580000 | 1.660000 | 65.800000 | 0.611000 | 289.000000 | 440.000000 | 1.038980 | 4.010000 | 2.000000 | 14.900000 | 9.000000 | 1.000000 |
Чтобы стало нагляднее, можем использовать DataFrame.hist 🛠️[doc] прямо из pandas:
import matplotlib.pyplot as plt
dataset.hist(figsize=(20, 10), layout=(-1, 5))
plt.show()
Видим, что у нас присутствует дисбаланс классов (распределение целевого признака is_red). Почти все распределения признаков одномодальные. Можем оценить диапазон значений признаков.
import seaborn as sns
plt.figure(figsize=(8, 8))
heatmap = sns.heatmap(
dataset.corr(method="pearson", numeric_only=True).round(2),
annot=True,
square=True,
cmap="RdBu_r",
vmax=1,
vmin=-1,
cbar_kws={"fraction": 0.01}, # shrink colour bar
linewidth=2,
)
heatmap.set_xticklabels(
heatmap.get_xticklabels(), rotation=45, horizontalalignment="right"
)
heatmap.set_title("Correalation heatmap", fontdict={"fontsize": 18}, pad=16)
plt.show()
Видим, что два признака имеют высокую корреляцию между собой (free_sulfur_dioxide и total_sulfur_dioxide). Почему это плохо и что с этим делать ✏️[blog].
Также важно отметить, что корреляция Пирсона, посчитанная для категориальных признаков, может давать некорректные и бессмысленные результаты.
Поэтому на практике хорошо себя показывает Phi_K 🛠️[doc] ($\phi_k$) корреляция. Она, в отличие от корреляции Пирсона, учитывает нелинейные зависимости и работает с категориальными признаками.
!pip install -q phik
━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━ 686.1/686.1 kB 3.9 MB/s eta 0:00:00
В этот раз отсортируем коэффициенты корреляции по целевой переменной:
import phik
plt.figure(figsize=(8, 8))
sotred_columns = (
dataset.phik_matrix(interval_cols=dataset.columns)
.round(2)
.sort_values("is_red", ascending=False, axis=1)
.columns
)
heatmap = sns.heatmap(
dataset.phik_matrix(interval_cols=dataset.columns)
.round(2)
.sort_values("is_red", ascending=False, axis=1)
.reindex(sotred_columns),
annot=True,
square=True,
cmap="Blues",
vmax=1,
vmin=0,
cbar_kws={"fraction": 0.01}, # shrink colour bar
linewidth=2,
)
heatmap.set_xticklabels(
heatmap.get_xticklabels(), rotation=45, horizontalalignment="right"
)
heatmap.set_title("Phik heatmap sorted by target", fontdict={"fontsize": 18}, pad=16)
plt.show()
Можем выделить признаки, которые имеют наибольший коэффициент. Посмотрим как они взаимодействуют друг с другом.
При большом количестве объектов бывает сложно понять, какие есть зависимости (объекты накладываются друг на друга), поэтому мы возьмем случайные 1000 объектов из нашего датасета, чтобы было нагляднее. Воспользуемся seaborn.pairplot 🛠️[doc].
sns.set_style("whitegrid")
sns.pairplot(
dataset.sample(1000),
vars=["total_sulfur_dioxide", "volatile_acidity", "chlorides"],
hue="is_red",
)
plt.show()
Можем отметить, что признак total_sulfur_dioxide действительно хорошо разделяется по целевому признаку. Также хорошее разделение видим в паре признаков total_sulfur_dioxide и volatile_acidity.
В датасете есть категориальный признак quality. Посмотрим, как распределен total_sulfur_dioxide в разрезе по целевому признаку и по каждому значению quality:
sns.catplot(
data=dataset,
x="quality",
y="total_sulfur_dioxide",
hue="is_red",
aspect=2,
alpha=0.5,
kind="violin", # strip, swarm, box, violin, boxen, point, bar, or count
split=True,
)
plt.show()
Видим, что в 9 категории нет объектов другого класса, но такой график не отражает количество объектов в категориях. Можем это посмотреть, воспользовавшись pandas.crosstab 🛠️[doc]:
cross_tab = sns.heatmap(
pd.crosstab(dataset["is_red"], dataset["quality"], normalize=True).round(4),
annot=True,
square=True,
cmap="Blues",
cbar_kws={"fraction": 0.01},
linewidth=1,
)
cross_tab.set_title("Crosstab heatmap", fontdict={"fontsize": 18}, pad=16)
plt.show()
Теперь видно, что объектов 9 категории довольно мало. Также можем оценить, в каких категориях сконцетрированы объекты.
Еще один полезный тип графика — это seaborn.relplot 🛠️[doc]. Eго удобно использовать, если у нас есть временной признак, тогда мы можем построить зависимость признака относительно времени.
У нас такого признака нет, построим два признака с раскраской по категориальной переменной quality:
relplot = sns.relplot(
data=dataset,
x="residual_sugar",
y="alcohol",
hue="quality",
kind="scatter", # or line
aspect=2,
alpha=0.6,
palette="Set2",
)
relplot.set_xticklabels(horizontalalignment="right", step=2)
plt.show()
Видно, что объекты 5 категории в среднем находятся ниже 7 категории, построим тот же seaborn.relplot, но уже для двух категорий. В этот раз для примера будем использовать параметр kind="line", который отвечает за вид графика:
relplot = sns.relplot(
data=dataset.query("quality == (5,7)"),
x="residual_sugar",
y="alcohol",
hue="quality",
kind="line",
aspect=2,
alpha=0.3,
palette="Set2",
)
relplot.set_xticklabels(horizontalalignment="right", step=2)
plt.show()
Итоги:
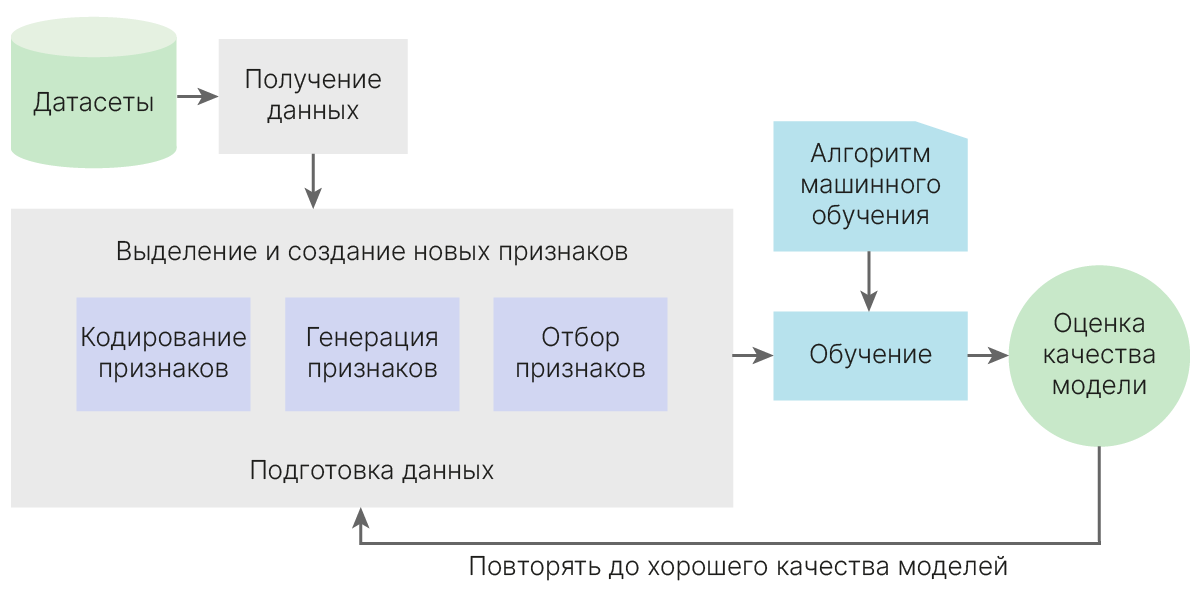
Общая схема классического машинного обучения выглядит так. Даже в случае нейросетей некоторая предобработка исходных данных все равно не бывает лишней.
Генерация признаков — процесс создания числовых представлений данных.
Процесс создания признаков зависит от модели, которую мы собираемся использовать.
Для одних моделей (линейные, древесные модели) может быть полезно добавить признаки, полученные делением/перемножением исходных. Другие модели (например, SVM) могут провести эти операции сами, причем, экономнее/менее переобучаясь.
Добавление признаков, явно зависящих друг от друга, мешает некоторым моделям. Например, плохая идея — дублировать в линейной модели признаки X1 и X2, или добавлять их сумму.

Feature engineering является критическим местом (bottleneck), в машинном обучении. На этом этапе определяется, что модель будет знать о данных. Можно упростить задачу для модели, подав подходящий набор признаков, или ввести модель в заблуждения, допустив критические ошибки.
ML модели “ленивы”: если дать модели подсказку об ответе, она не будет искать сложные закономерности в данных. Такой подсказкой может быть id покупателя. Для покупателя, который каждую неделю покупает одно и то же, модель выучит, что надо предсказывать все по признаку id. Если покупатель изменит свое поведение или придет новый покупатель, то модель может выдавать неадекватные предсказания. Такую же роль может сыграть информация о номере эксперимента, лаборатории, в которой его проводили, аспиранте, который его проводил, и т.д. Эта ситуация будет называться Data Leakage. Перечисленная выше информация может быть важна для анализа исходных данных, например, можно выяснить, что у прибора в лаборатории А проблемы с калибровкой.
При решении задачи машинного обучения важно оценивать результат. Один из способов оценить результат — это сделать базовое решение задачи. Как правило, это решение, которое требует мало времени для реализации и явно лучше случайного ответа. Далее, улучшая решение, мы можем понимать, насколько оно лучше базового и что на это повлияло (подбор гиперпараметров, кодирование признаков, генерация признаков и многое другое).
Постановка задачи следующая:
Автомобильная компания выходит на новый рынок. После интенсивного исследования рынка они пришли к выводу, что поведение нового рынка похоже на их существующий рынок. На существующем рынке отдел продаж, изучив поведение клиентов, разделил их на сегменты (классы) A, B, C, D. Для нового рынка данных о поведении нет, но есть данные о потенциальных клиентах (пол, возраст, образование, уровень дохода, состав семьи и т.д.). Нужно классифицировать потенциальных клиентов.
Загрузим датасет Customer-Segmentation 🛠️[doc] и посмотрим, какие есть признаки:
import pandas as pd
data = pd.read_csv(
"https://edunet.kea.su/repo/EduNet-web_dependencies/datasets/customer_segmentation.csv"
)
data
| ID | Gender | Ever_Married | Age | Graduated | Profession | Work_Experience | Spending_Score | Family_Size | Var_1 | Segmentation | |
|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 462809 | Male | No | 22 | No | Healthcare | 1.0 | Low | 4.0 | Cat_4 | D |
| 1 | 462643 | Female | Yes | 38 | Yes | Engineer | NaN | Average | 3.0 | Cat_4 | A |
| 2 | 466315 | Female | Yes | 67 | Yes | Engineer | 1.0 | Low | 1.0 | Cat_6 | B |
| 3 | 461735 | Male | Yes | 67 | Yes | Lawyer | 0.0 | High | 2.0 | Cat_6 | B |
| 4 | 462669 | Female | Yes | 40 | Yes | Entertainment | NaN | High | 6.0 | Cat_6 | A |
| ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... |
| 8063 | 464018 | Male | No | 22 | No | NaN | 0.0 | Low | 7.0 | Cat_1 | D |
| 8064 | 464685 | Male | No | 35 | No | Executive | 3.0 | Low | 4.0 | Cat_4 | D |
| 8065 | 465406 | Female | No | 33 | Yes | Healthcare | 1.0 | Low | 1.0 | Cat_6 | D |
| 8066 | 467299 | Female | No | 27 | Yes | Healthcare | 1.0 | Low | 4.0 | Cat_6 | B |
| 8067 | 461879 | Male | Yes | 37 | Yes | Executive | 0.0 | Average | 3.0 | Cat_4 | B |
8068 rows × 11 columns
data.info()
<class 'pandas.core.frame.DataFrame'> RangeIndex: 8068 entries, 0 to 8067 Data columns (total 11 columns): # Column Non-Null Count Dtype --- ------ -------------- ----- 0 ID 8068 non-null int64 1 Gender 8068 non-null object 2 Ever_Married 7928 non-null object 3 Age 8068 non-null int64 4 Graduated 7990 non-null object 5 Profession 7944 non-null object 6 Work_Experience 7239 non-null float64 7 Spending_Score 8068 non-null object 8 Family_Size 7733 non-null float64 9 Var_1 7992 non-null object 10 Segmentation 8068 non-null object dtypes: float64(2), int64(2), object(7) memory usage: 693.5+ KB
Посмотрим признак Family_Size. По смыслу он не подразумевает вещественных значений, хотя хранится в формате float64:
data["Family_Size"].unique()
array([ 4., 3., 1., 2., 6., nan, 5., 8., 7., 9.])
Действительно, все значения целые. В нашем случае не критично, но если датасет большой, то имеет смысл привести формат данных к более компактному представлению.
Видно, что есть пропуски. Для базового решения можно удалить или заполнить нулями. Мы заполним нулями и переведем все категориальные признаки в формат one-hot encoding
from sklearn.model_selection import train_test_split
data["Segmentation"] = data["Segmentation"].map({"D": 1, "A": 2, "C": 3, "B": 4})
data.fillna(0, inplace=True)
data.drop(columns=["ID"], inplace=True)
data = pd.get_dummies(data) # to ohe
x = data.drop(columns=["Segmentation"], axis=1)
y = data["Segmentation"]
x_train, x_test, y_train, y_test = train_test_split(
x, y, test_size=0.3, random_state=42
)
print("Shape of x_train :", x_train.shape)
print("Shape of y_train :", y_train.shape)
print("Shape of x_test :", x_test.shape)
print("Shape of y_test :", y_test.shape)
Shape of x_train : (5647, 32) Shape of y_train : (5647,) Shape of x_test : (2421, 32) Shape of y_test : (2421,)
Запустим модель случайного леса и посмотрим, какой результат получится:
import seaborn as sns
import matplotlib.pyplot as plt
from sklearn.ensemble import RandomForestClassifier
from sklearn.metrics import confusion_matrix
from sklearn.metrics import classification_report
def train_and_plot(x_train, y_train, x_test, y_test):
model = RandomForestClassifier(random_state=42)
model.fit(x_train, y_train)
y_pred = model.predict(x_test)
# evaluating the model
print("Training Accuracy: ", model.score(x_train, y_train).round(3))
print("Testing Accuracy: ", model.score(x_test, y_test).round(3))
# confusion matrix
cm = confusion_matrix(y_test, y_pred, normalize="all")
plt.figure(figsize=(6, 6))
sns.heatmap(cm, annot=True)
# classification report
cr = classification_report(y_test, y_pred)
print(cr)
train_and_plot(x_train, y_train, x_test, y_test)
Training Accuracy: 0.966
Testing Accuracy: 0.491
precision recall f1-score support
1 0.62 0.71 0.66 692
2 0.41 0.38 0.39 597
3 0.50 0.49 0.49 580
4 0.37 0.35 0.36 552
accuracy 0.49 2421
macro avg 0.48 0.48 0.48 2421
weighted avg 0.48 0.49 0.49 2421
Мы можем использовать другие модели, чтобы получить новые признаки для наших данных. Например, используя разные модели, можем использовать их предсказания, как новые признаки. В каком-то смысле мы это уже делали на прошлой лекции, разбирая блендинг.
Генерация бинарного признакового пространства с помощью RandomForest:
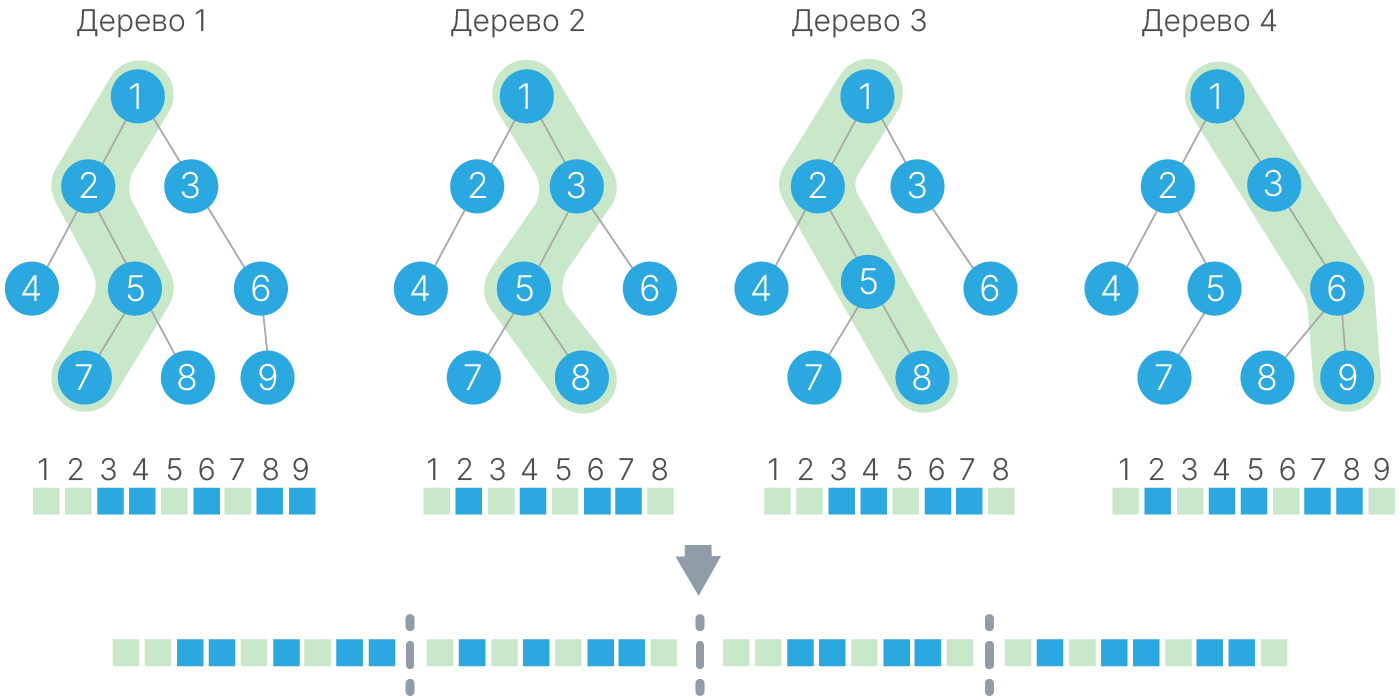
Добавим новый бинарный признак, используя IsolationForest:
from sklearn.ensemble import IsolationForest
n_estimators = 300
contamination = 0.1 # threshold
model = IsolationForest(
n_estimators=n_estimators, contamination="auto", random_state=42
)
model.fit(x_train)
x_train["new_feature_1"] = model.predict(x_train)
x_test["new_feature_1"] = model.predict(x_test)
Обратите внимание, что при генерации новых признаков важно не переобучиться и не допустить утечку данных, поэтому мы обучаем IsolationForest на трейне, а не на всей выборке.
train_and_plot(x_train, y_train, x_test, y_test)
Training Accuracy: 0.966
Testing Accuracy: 0.494
precision recall f1-score support
1 0.62 0.71 0.66 692
2 0.42 0.39 0.40 597
3 0.51 0.49 0.50 580
4 0.36 0.34 0.35 552
accuracy 0.49 2421
macro avg 0.48 0.48 0.48 2421
weighted avg 0.49 0.49 0.49 2421
Качество немного улучшилось.
Можем использовать алгоритмы кластеризации, чтобы получить новые признаки. Количество кластеров можно подбирать, оно не всегда должно быть равно количеству классов. Для примера возьмем n_clusters=12.
import sklearn.cluster as cluster
clusters = cluster.KMeans(n_clusters=12, n_init="auto", random_state=42).fit(x_train)
x_train["new_feature_2"] = clusters.predict(x_train)
x_test["new_feature_2"] = clusters.predict(x_test)
x_train.head(3)
| Age | Work_Experience | Family_Size | Gender_Female | Gender_Male | Ever_Married_0 | Ever_Married_No | Ever_Married_Yes | Graduated_0 | Graduated_No | ... | Var_1_0 | Var_1_Cat_1 | Var_1_Cat_2 | Var_1_Cat_3 | Var_1_Cat_4 | Var_1_Cat_5 | Var_1_Cat_6 | Var_1_Cat_7 | new_feature_1 | new_feature_2 | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 5015 | 49 | 1.0 | 2.0 | False | True | False | False | True | False | False | ... | False | False | False | False | False | False | True | False | 1 | 6 |
| 5755 | 87 | 0.0 | 2.0 | False | True | False | False | True | False | False | ... | False | False | False | False | False | False | True | False | 1 | 4 |
| 7781 | 28 | 8.0 | 3.0 | True | False | False | True | False | False | False | ... | False | False | False | False | False | False | True | False | 1 | 2 |
3 rows × 34 columns
train_and_plot(x_train, y_train, x_test, y_test)
Training Accuracy: 0.966
Testing Accuracy: 0.503
precision recall f1-score support
1 0.63 0.71 0.67 692
2 0.43 0.40 0.42 597
3 0.50 0.49 0.50 580
4 0.39 0.37 0.38 552
accuracy 0.50 2421
macro avg 0.49 0.49 0.49 2421
weighted avg 0.50 0.50 0.50 2421
Можем генерировать признаки самостоятельно, придумывая различные гипотезы и эвристики. Это творческий процесс, и нет точного рецепта, какие признаки стоит сгенерировать. Также генерация признаков зависит от модели, для которой эти признаки генерируем. Например, признаки, описывающие нелинейное взаимодействие, могут кардинально изменить качество линейной модели.
Наша задача — рассмотреть инструменты для генерации признаков.
data = pd.read_csv(
"https://edunet.kea.su/repo/EduNet-web_dependencies/datasets/customer_segmentation.csv"
)
data.dropna(inplace=True)
data.head(3)
| ID | Gender | Ever_Married | Age | Graduated | Profession | Work_Experience | Spending_Score | Family_Size | Var_1 | Segmentation | |
|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 462809 | Male | No | 22 | No | Healthcare | 1.0 | Low | 4.0 | Cat_4 | D |
| 2 | 466315 | Female | Yes | 67 | Yes | Engineer | 1.0 | Low | 1.0 | Cat_6 | B |
| 3 | 461735 | Male | Yes | 67 | Yes | Lawyer | 0.0 | High | 2.0 | Cat_6 | B |
В этот раз закодируем признаки по их присутствию в данных:
data["Profession"].value_counts()
Profession Artist 2192 Healthcare 1077 Entertainment 809 Doctor 592 Engineer 582 Executive 505 Lawyer 500 Marketing 233 Homemaker 175 Name: count, dtype: int64
data["Gender"] = data["Gender"].map({"Male": 1, "Female": 0})
data["Graduated"] = data["Graduated"].map({"Yes": 1, "No": 0})
data["Ever_Married"] = data["Ever_Married"].map({"Yes": 1, "No": 0})
data["Spending_Score"] = data["Spending_Score"].map({"High": 1, "Average": 2, "Low": 3})
data["Var_1"] = data["Var_1"].map(
{"Cat_6": 1, "Cat_4": 2, "Cat_3": 3, "Cat_2": 4, "Cat_7": 5, "Cat_1": 6, "Cat_5": 7}
)
data["Segmentation"] = data["Segmentation"].map({"D": 1, "A": 2, "C": 3, "B": 4})
data["Profession"] = data["Profession"].map(
{
"Artist": 1,
"Healthcare": 2,
"Entertainment": 3,
"Doctor": 4,
"Engineer": 5,
"Executive": 5,
"Lawyer": 6,
"Marketing": 7,
"Homemaker": 8,
}
)
data.head(5)
| ID | Gender | Ever_Married | Age | Graduated | Profession | Work_Experience | Spending_Score | Family_Size | Var_1 | Segmentation | |
|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 462809 | 1 | 0 | 22 | 0 | 2 | 1.0 | 3 | 4.0 | 2 | 1 |
| 2 | 466315 | 0 | 1 | 67 | 1 | 5 | 1.0 | 3 | 1.0 | 1 | 4 |
| 3 | 461735 | 1 | 1 | 67 | 1 | 6 | 0.0 | 1 | 2.0 | 1 | 4 |
| 5 | 461319 | 1 | 1 | 56 | 0 | 1 | 0.0 | 2 | 2.0 | 1 | 3 |
| 6 | 460156 | 1 | 0 | 32 | 1 | 2 | 1.0 | 3 | 3.0 | 1 | 3 |
Можем производить различные математические операции с признаками: перемножать, делить, складывать и т.д. Для временных рядов полезно брать производные, считать стандартное отклонение и т.д
Для примера перемножим признак Age и Work_Experience
data["new_feature_1"] = data["Age"] * data["Work_Experience"]
data.head(3)
| ID | Gender | Ever_Married | Age | Graduated | Profession | Work_Experience | Spending_Score | Family_Size | Var_1 | Segmentation | new_feature_1 | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 462809 | 1 | 0 | 22 | 0 | 2 | 1.0 | 3 | 4.0 | 2 | 1 | 22.0 |
| 2 | 466315 | 0 | 1 | 67 | 1 | 5 | 1.0 | 3 | 1.0 | 1 | 4 | 67.0 |
| 3 | 461735 | 1 | 1 | 67 | 1 | 6 | 0.0 | 1 | 2.0 | 1 | 4 | 0.0 |
Полезный метод для генерации признаков — это pd.groupby 🛠️[doc].
Посчитаем средний возраст для каждой профессии и опыта работы:
data["new_feature_2"] = data.groupby(["Profession", "Work_Experience"], as_index=False)[
"Age"
].transform("mean")
data.head(3)
| ID | Gender | Ever_Married | Age | Graduated | Profession | Work_Experience | Spending_Score | Family_Size | Var_1 | Segmentation | new_feature_1 | new_feature_2 | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 462809 | 1 | 0 | 22 | 0 | 2 | 1.0 | 3 | 4.0 | 2 | 1 | 22.0 | 26.739255 |
| 2 | 466315 | 0 | 1 | 67 | 1 | 5 | 1.0 | 3 | 1.0 | 1 | 4 | 67.0 | 48.608466 |
| 3 | 461735 | 1 | 1 | 67 | 1 | 6 | 0.0 | 1 | 2.0 | 1 | 4 | 0.0 | 75.520548 |
Можно использовать разные функции. Например, медиану:
data["new_feature_3"] = data.groupby(["Spending_Score", "Family_Size"], as_index=False)[
"Var_1"
].transform("median")
data.head(3)
| ID | Gender | Ever_Married | Age | Graduated | Profession | Work_Experience | Spending_Score | Family_Size | Var_1 | Segmentation | new_feature_1 | new_feature_2 | new_feature_3 | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 462809 | 1 | 0 | 22 | 0 | 2 | 1.0 | 3 | 4.0 | 2 | 1 | 22.0 | 26.739255 | 1.0 |
| 2 | 466315 | 0 | 1 | 67 | 1 | 5 | 1.0 | 3 | 1.0 | 1 | 4 | 67.0 | 48.608466 | 1.0 |
| 3 | 461735 | 1 | 1 | 67 | 1 | 6 | 0.0 | 1 | 2.0 | 1 | 4 | 0.0 | 75.520548 | 1.0 |
Также можем написать свою функции и использовать ее:
import numpy as np
custom_func = lambda x: np.percentile(x, 75)
data["new_feature_4"] = data.groupby(["Spending_Score", "Family_Size"], as_index=False)[
"Age"
].transform(custom_func)
data.head(3)
| ID | Gender | Ever_Married | Age | Graduated | Profession | Work_Experience | Spending_Score | Family_Size | Var_1 | Segmentation | new_feature_1 | new_feature_2 | new_feature_3 | new_feature_4 | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 462809 | 1 | 0 | 22 | 0 | 2 | 1.0 | 3 | 4.0 | 2 | 1 | 22.0 | 26.739255 | 1.0 | 32.0 |
| 2 | 466315 | 0 | 1 | 67 | 1 | 5 | 1.0 | 3 | 1.0 | 1 | 4 | 67.0 | 48.608466 | 1.0 | 57.0 |
| 3 | 461735 | 1 | 1 | 67 | 1 | 6 | 0.0 | 1 | 2.0 | 1 | 4 | 0.0 | 75.520548 | 1.0 | 79.0 |
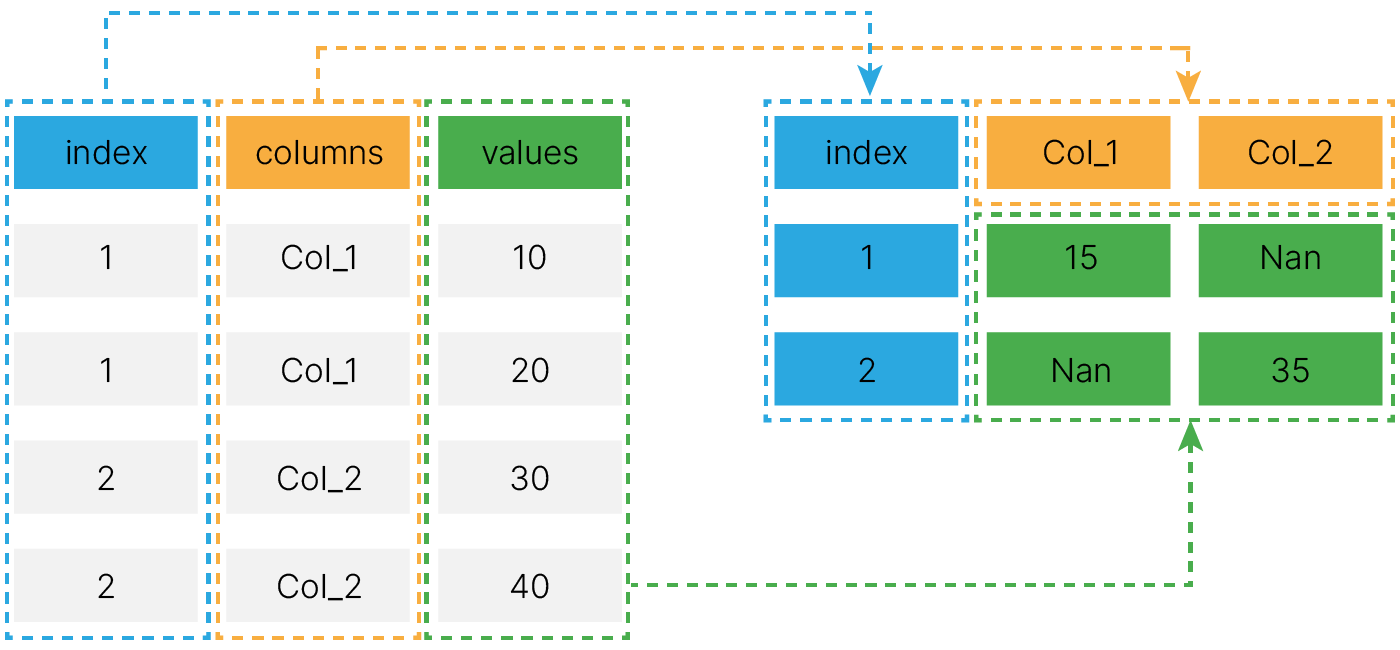
Можем сгенерировать новую таблицу признаков, используя pivot_table 🛠️[doc]:
data.pivot_table(
index="index",
columns=["columns"],
values=["values"],
aggfunc=["mean"],
)
data_pivot = data.pivot_table(
index="Family_Size", # indexes
columns=["Var_1"], # new columns
values=["Age"], # values are used fo aggfunc
aggfunc=["mean", "count"], # mean/count/sum
).fillna(0)
data_pivot.columns = [f"Cat{int(i[2])}_{i[0]}" for i in data_pivot.columns]
data_pivot.reset_index(inplace=True)
data_pivot.head(3)
| Family_Size | Cat1_mean | Cat2_mean | Cat3_mean | Cat4_mean | Cat5_mean | Cat6_mean | Cat7_mean | Cat1_count | Cat2_count | Cat3_count | Cat4_count | Cat5_count | Cat6_count | Cat7_count | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 1.0 | 49.434354 | 44.256637 | 46.272000 | 41.333333 | 40.041667 | 44.230769 | 42.777778 | 914.0 | 113.0 | 125.0 | 45.0 | 24.0 | 13.0 | 9.0 |
| 1 | 2.0 | 51.064619 | 43.015625 | 39.494048 | 45.171053 | 44.159091 | 42.187500 | 40.888889 | 1563.0 | 192.0 | 168.0 | 76.0 | 44.0 | 32.0 | 18.0 |
| 2 | 3.0 | 42.033257 | 38.161290 | 36.627907 | 36.263889 | 37.935484 | 34.476190 | 36.333333 | 872.0 | 155.0 | 129.0 | 72.0 | 31.0 | 21.0 | 12.0 |
А потом соединить ее с исходной:
data = data.merge(data_pivot, how="inner", on="Family_Size")
data.head(3)
| ID | Gender | Ever_Married | Age | Graduated | Profession | Work_Experience | Spending_Score | Family_Size | Var_1 | ... | Cat5_mean | Cat6_mean | Cat7_mean | Cat1_count | Cat2_count | Cat3_count | Cat4_count | Cat5_count | Cat6_count | Cat7_count | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 462809 | 1 | 0 | 22 | 0 | 2 | 1.0 | 3 | 4.0 | 2 | ... | 37.114286 | 39.047619 | 39.230769 | 726.0 | 158.0 | 132.0 | 89.0 | 35.0 | 21.0 | 13.0 |
| 1 | 465176 | 0 | 1 | 55 | 1 | 1 | 1.0 | 2 | 4.0 | 1 | ... | 37.114286 | 39.047619 | 39.230769 | 726.0 | 158.0 | 132.0 | 89.0 | 35.0 | 21.0 | 13.0 |
| 2 | 464942 | 1 | 0 | 19 | 0 | 2 | 4.0 | 3 | 4.0 | 2 | ... | 37.114286 | 39.047619 | 39.230769 | 726.0 | 158.0 | 132.0 | 89.0 | 35.0 | 21.0 | 13.0 |
3 rows × 29 columns
Мы довольно быстро увеличили количество признаков в нашем датасете. Проверим, как это повлияло на качество:
data.drop(columns=["ID"], inplace=True)
x = data.drop(columns=["Segmentation"], axis=1)
y = data["Segmentation"]
x_train, x_test, y_train, y_test = train_test_split(
x, y, test_size=0.3, random_state=42
)
print("Shape of x_train :", x_train.shape)
print("Shape of y_train :", y_train.shape)
print("Shape of x_test :", x_test.shape)
print("Shape of y_test :", y_test.shape)
Shape of x_train : (4665, 27) Shape of y_train : (4665,) Shape of x_test : (2000, 27) Shape of y_test : (2000,)
train_and_plot(x_train, y_train, x_test, y_test)
Training Accuracy: 0.964
Testing Accuracy: 0.472
precision recall f1-score support
1 0.60 0.67 0.63 530
2 0.38 0.36 0.37 500
3 0.50 0.52 0.51 495
4 0.35 0.32 0.34 475
accuracy 0.47 2000
macro avg 0.46 0.47 0.46 2000
weighted avg 0.46 0.47 0.47 2000
Стало хуже. Возможно, какие-то признаки и улучшали качество, но другие его испортили сильнее. После того, как мы нагенерировали много признаков, стоит задуматься о том, какие признаки улучшают качество, а какие его ухудшают.
Рассмотрим способы и подходы, чтобы отделять полезные признаки от бесполезных.
Не всегда нужно использовать все признаки, которые у нас есть и которые мы можем нагенерировать. Нам нужны только те признаки, которые позволяют решить задачу.
Признаки могут коррелировать с целевой переменной случайно. Если провести эксперимент и сгенерировать большое количество случайных признаков для нашего датасета, мы можем заметить эффект, что некоторые из них действительно улучшают качество предсказания, но это никак не связано с реальной закономерностью. На новых (тестовых) данных мы обнаружим неадекватность предсказаний.
В более общем случае можно говорить, что в многомерном пространстве почти всегда можно найти корреляции:
Скорость работы модели часто имеет значение
В практических задачах часто приходится искать компромисс между точностью предсказания модели и временем его получения.
Спектр таких задач достаточно широк: от проблем построения быстрых систем ранжирования рекламных объявлений в интернет-маркетинге до построения быстрых систем распознавания сложных событий на ускорителях заряженных частиц.
Вычислительная сложность модели растёт с увеличением числа входных признаков, поэтому работающие с меньшим числом признаков модели являются предпочтительными.
Можно попытаться перебрать все возможные комбинации признаков. Однако даже для 100 признаков такой подход будет считаться до конца Вселенной.
Потому прибегают к эвристикам, которые могут пропускать оптимальное решение.
Самый простой подход к отбору признаков — это одномерный подход. В нём оценивается связь каждого признака с целевой переменной, например, измеряется корреляция.
Такой подход не учитывает сложные закономерности: в нём все признаки считаются независимыми, тогда как в машинном обучении модели учитывают взаимное влияние признаков, их пар или даже более сложные взаимодействия.
Будем использовать датасет Титаник, загрузим данные:
import pandas as pd
dataset = pd.read_csv(
"https://edunet.kea.su/repo/EduNet-web_dependencies/datasets/titanic.csv"
)
dataset.head(5)
| PassengerId | Survived | Pclass | Name | Sex | Age | SibSp | Parch | Ticket | Fare | Cabin | Embarked | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 1 | 0 | 3 | Braund, Mr. Owen Harris | male | 22.0 | 1 | 0 | A/5 21171 | 7.2500 | NaN | S |
| 1 | 2 | 1 | 1 | Cumings, Mrs. John Bradley (Florence Briggs Th... | female | 38.0 | 1 | 0 | PC 17599 | 71.2833 | C85 | C |
| 2 | 3 | 1 | 3 | Heikkinen, Miss. Laina | female | 26.0 | 0 | 0 | STON/O2. 3101282 | 7.9250 | NaN | S |
| 3 | 4 | 1 | 1 | Futrelle, Mrs. Jacques Heath (Lily May Peel) | female | 35.0 | 1 | 0 | 113803 | 53.1000 | C123 | S |
| 4 | 5 | 0 | 3 | Allen, Mr. William Henry | male | 35.0 | 0 | 0 | 373450 | 8.0500 | NaN | S |
Сделаем предобработку и кодирование признаков:
import numpy as np
from sklearn.experimental import enable_iterative_imputer
from sklearn.impute import IterativeImputer
title_mapping = {"Mr": 4, "Miss": 3, "Mrs": 2, "Master": 1}
# get title from name and encode by title_mapping, another titles fill zero
dataset["Title"] = (
dataset.Name.str.extract(" ([A-Za-z]+)\.", expand=False)
.map(title_mapping)
.fillna(0)
)
# encode to binary column
dataset["Sex"] = dataset["Sex"].map({"male": 1, "female": 0})
# ordinary encode by values_count
dataset["Embarked"] = dataset["Embarked"].map({np.nan: 0, "S": 0, "Q": 1, "C": 2})
# parse cabin to get deck A, B, C etc
dataset["Deck"] = dataset["Cabin"].str.extract("([A-Za-z])")
# parse cabin to get cabin number
dataset["Cabin_Number"] = dataset["Cabin"].str.extract("(\d+)").astype(float).fillna(0)
# get sqrt fare
dataset["sqrt_Fare"] = np.sqrt(dataset["Fare"])
# deck encode to ohe format
deck_to_ohe = pd.get_dummies(dataset["Deck"], prefix="Cab", dummy_na=True)
# add ohe features to dataset
dataset = pd.concat([dataset, deck_to_ohe], axis=1)
# drop unnecessary columns
dataset.drop(
columns=["Name", "Ticket", "Cabin", "PassengerId", "Deck", "Fare"], inplace=True
)
# train imputer to fiil nans for age
imputer = IterativeImputer()
imputer.fit(dataset[["Age", "Title", "sqrt_Fare"]])
# fill age nans by imputer
dataset["Age"] = imputer.transform(dataset[["Age", "Title", "sqrt_Fare"]])
dataset.head(5)
| Survived | Pclass | Sex | Age | SibSp | Parch | Embarked | Title | Cabin_Number | sqrt_Fare | Cab_A | Cab_B | Cab_C | Cab_D | Cab_E | Cab_F | Cab_G | Cab_T | Cab_nan | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 0 | 3 | 1 | 22.0 | 1 | 0 | 0 | 4.0 | 0.0 | 2.692582 | False | False | False | False | False | False | False | False | True |
| 1 | 1 | 1 | 0 | 38.0 | 1 | 0 | 2 | 2.0 | 85.0 | 8.442944 | False | False | True | False | False | False | False | False | False |
| 2 | 1 | 3 | 0 | 26.0 | 0 | 0 | 0 | 3.0 | 0.0 | 2.815138 | False | False | False | False | False | False | False | False | True |
| 3 | 1 | 1 | 0 | 35.0 | 1 | 0 | 0 | 2.0 | 123.0 | 7.286975 | False | False | True | False | False | False | False | False | False |
| 4 | 0 | 3 | 1 | 35.0 | 0 | 0 | 0 | 4.0 | 0.0 | 2.837252 | False | False | False | False | False | False | False | False | True |
Дополнительно добавим в наш датасет случайные признаки, чтобы оценить, как методы отбора будут учитывать их:
random_features = pd.read_csv(
"https://edunet.kea.su/repo/EduNet-web_dependencies/datasets/random_features.csv"
)
dataset = pd.concat([dataset, random_features], axis=1)
dataset.head(5)
| Survived | Pclass | Sex | Age | SibSp | Parch | Embarked | Title | Cabin_Number | sqrt_Fare | ... | Cab_E | Cab_F | Cab_G | Cab_T | Cab_nan | random_1 | random_2 | random_3 | random_4 | random_5 | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 0 | 3 | 1 | 22.0 | 1 | 0 | 0 | 4.0 | 0.0 | 2.692582 | ... | False | False | False | False | True | 9.4 | 3 | 1.0 | 2 | 2 |
| 1 | 1 | 1 | 0 | 38.0 | 1 | 0 | 2 | 2.0 | 85.0 | 8.442944 | ... | False | False | False | False | False | 9.8 | 2 | 1.0 | 1 | 1 |
| 2 | 1 | 3 | 0 | 26.0 | 0 | 0 | 0 | 3.0 | 0.0 | 2.815138 | ... | False | False | False | False | True | 9.8 | 3 | 4.0 | 2 | 2 |
| 3 | 1 | 1 | 0 | 35.0 | 1 | 0 | 0 | 2.0 | 123.0 | 7.286975 | ... | False | False | False | False | False | 9.8 | 2 | 1.0 | 3 | 1 |
| 4 | 0 | 3 | 1 | 35.0 | 0 | 0 | 0 | 4.0 | 0.0 | 2.837252 | ... | False | False | False | False | True | 9.4 | 3 | 3.0 | 2 | 1 |
5 rows × 24 columns
Оценим, как признаки коррелируют с целевой переменной. Будем использовать корреляцию Пирсона, но можно использовать и другую:
import matplotlib.pyplot as plt
from scipy import stats
from sklearn.model_selection import train_test_split
x = dataset.drop(columns=["Survived"])
y = dataset["Survived"]
x_train, x_test, y_train, y_test = train_test_split(
x, y, test_size=0.2, random_state=42
)
correlations = [] # create a storage for correlations
for column in x:
r, p_value = stats.pearsonr(x_train[column], y_train) # compute Pearson and R
correlations.append((column, abs(r).round(3))) # add to storage
df_corr = (
pd.DataFrame(correlations, columns=["Corr_feature", "Correlation"])
.sort_values("Correlation", ascending=False)
.reset_index(drop=True)
)
sorted_idx = np.argsort(df_corr["Correlation"])
fig = plt.figure(figsize=(10, 5))
plt.barh(
range(df_corr.shape[0]), df_corr["Correlation"].iloc[sorted_idx], align="center"
)
plt.yticks(range(df_corr.shape[0]), df_corr["Corr_feature"].iloc[sorted_idx])
plt.title("Feature Correlation")
plt.show()
Следующая идея — посчитать ROC-AUC по признаку, учитывая его как предсказание модели. Если ROC-AUC высокий (нас интересуют только абсолютные значения), то признак важный.
from sklearn.metrics import roc_auc_score
rocs = [] # create a storage for ROCs
for column in x:
# use feature as score directly
r1 = roc_auc_score(y_score=x_train[column], y_true=y_train)
# use feature as score in reversed manner
r2 = roc_auc_score(y_score=-x_train[column], y_true=y_train)
r = max(r1, r2)
rocs.append((column, r.round(3)))
df_rocs = (
pd.DataFrame(rocs, columns=["Rocs_feature", "Rocs"])
.sort_values("Rocs", ascending=False)
.reset_index(drop=True)
) # sort from highest to lowest
sorted_idx = np.argsort(df_rocs["Rocs"])
fig = plt.figure(figsize=(10, 5))
plt.barh(range(df_rocs.shape[0]), df_rocs["Rocs"].iloc[sorted_idx], align="center")
plt.yticks(range(df_rocs.shape[0]), df_rocs["Rocs_feature"].iloc[sorted_idx])
plt.title("Feature Rocs")
plt.show()
Или использовать уже нам знакомую phik корреляцию:
!pip install -q phik
import phik
df_phik = (
dataset.phik_matrix(interval_cols=dataset.columns)
.round(3)
.sort_values("Survived", ascending=False)
.reset_index()[["index", "Survived"]][1:]
.rename(columns={"index": "Phik_feature", "Survived": "Phik"})
.reset_index(drop=True)
)
sorted_idx = np.argsort(df_phik["Phik"])
fig = plt.figure(figsize=(10, 5))
plt.barh(range(df_phik.shape[0]), df_phik["Phik"].iloc[sorted_idx], align="center")
plt.yticks(range(df_phik.shape[0]), df_phik["Phik_feature"].iloc[sorted_idx])
plt.title("Feature Phik")
plt.show()
Посмотрим все методы в общей таблице, чтобы сравнить порядок признаков:
pd.concat([df_corr, df_rocs, df_phik], axis=1)
| Corr_feature | Correlation | Rocs_feature | Rocs | Phik_feature | Phik | |
|---|---|---|---|---|---|---|
| 0 | Sex | 0.542 | Title | 0.775 | Sex | 0.750 |
| 1 | Title | 0.426 | Sex | 0.766 | Cab_nan | 0.471 |
| 2 | Pclass | 0.322 | sqrt_Fare | 0.677 | Title | 0.462 |
| 3 | Cab_nan | 0.300 | Pclass | 0.674 | sqrt_Fare | 0.462 |
| 4 | sqrt_Fare | 0.296 | Cab_nan | 0.629 | random_1 | 0.363 |
| 5 | Cabin_Number | 0.228 | Cabin_Number | 0.627 | random_3 | 0.306 |
| 6 | Cab_B | 0.177 | Embarked | 0.567 | Cabin_Number | 0.306 |
| 7 | Embarked | 0.155 | Parch | 0.559 | Cab_B | 0.259 |
| 8 | Cab_E | 0.144 | random_1 | 0.550 | Cab_D | 0.219 |
| 9 | Cab_C | 0.127 | Cab_B | 0.541 | Cab_E | 0.211 |
| 10 | Cab_D | 0.123 | Cab_C | 0.534 | Age | 0.205 |
| 11 | Parch | 0.078 | SibSp | 0.534 | Pclass | 0.205 |
| 12 | random_1 | 0.062 | Cab_E | 0.527 | SibSp | 0.176 |
| 13 | random_3 | 0.059 | Age | 0.526 | Cab_C | 0.164 |
| 14 | Age | 0.058 | Cab_D | 0.520 | Parch | 0.147 |
| 15 | Cab_F | 0.056 | random_2 | 0.520 | Embarked | 0.099 |
| 16 | random_5 | 0.051 | random_5 | 0.520 | random_5 | 0.075 |
| 17 | SibSp | 0.048 | random_3 | 0.514 | Cab_F | 0.055 |
| 18 | random_2 | 0.034 | Cab_F | 0.507 | Cab_G | 0.000 |
| 19 | Cab_T | 0.029 | random_4 | 0.502 | Cab_T | 0.000 |
| 20 | Cab_A | 0.006 | Cab_A | 0.501 | Cab_A | 0.000 |
| 21 | Cab_G | 0.006 | Cab_T | 0.501 | random_2 | 0.000 |
| 22 | random_4 | 0.001 | Cab_G | 0.500 | random_4 | 0.000 |
Как мы видим, не все случайные признаки находятся в самом низу. Некоторые из них имеют более высокую оценку, чем наши исходные.
Важно! Это не означает, что признаки, которые находятся ниже случайных, плохие.
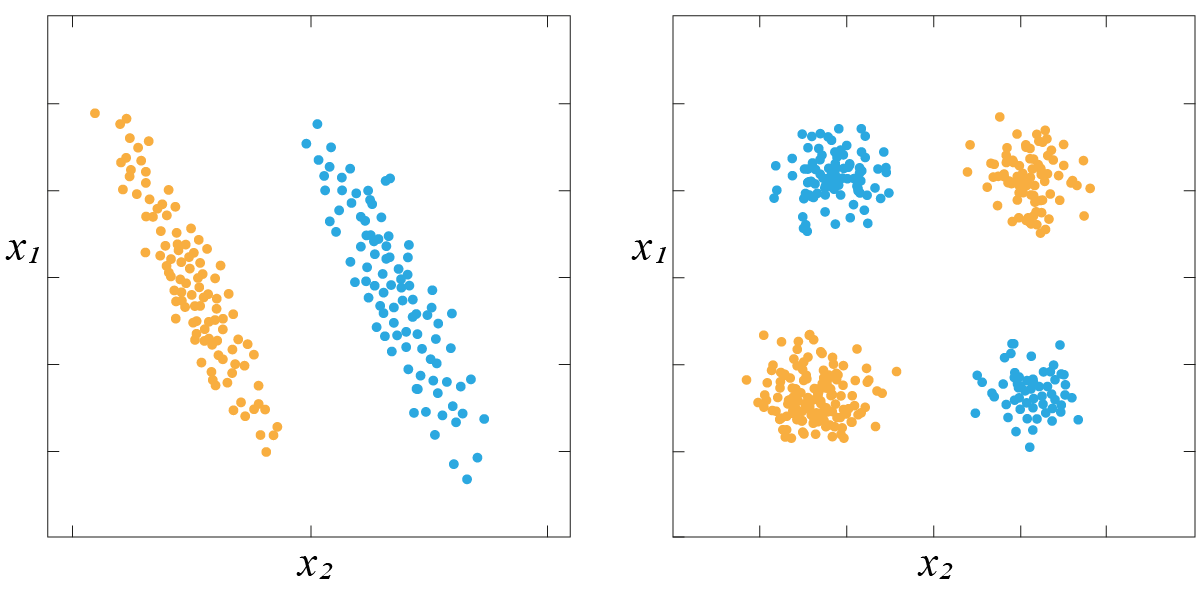
У подхода, при котором важности всех признаков оцениваются по отдельности, есть свои недостатки. На левом рисунке изображена двумерная выборка, для которой необходимо решить задачу классификации. Если спроецировать данную выборку на ось абсцисс, то она будет разделима, хотя и будут присутствовать ошибки. Если же спроецировать данную выборку на ось ординат, то все объекты разных классов перемешаются, и выборка будет неразделима. В этом случае при использовании любого метода одномерного оценивания информативности первый признак будет информативен, а второй — совершенно неинформативен.
Тем не менее, видно, что если использовать эти признаки одновременно, то классы будут разделимы идеально. Второй признак важен, но он важен только в совокупности с первым, и методы одномерного оценивания информативности не способны это определить.
На рисунке справа показана выборка, на которой одномерные методы оценки информативности работают ещё хуже. В этом случае, если спроецировать выборку на ось абсцисс или ординат, то объекты классов перемешаются, и в обоих случаях данные будут совершенно неразделимы. И, согласно любому из описанных методов, оба признака неинформативны. Тем не менее, если использовать их одновременно, то, например, решающее дерево может идеально решить данную задачу классификации.
Пример: влияние роста и веса при предсказании вероятности сердечного заболевания. Избыточный вес может являться важным фактором, но оценить, является ли он избыточным или нормальным, можно только зная рост пациента.
Использование весов признаков.
В линейных моделях перед признаками стоят веса. Веса при признаках можно интерпретировать как информативности: чем больше по модулю вес при признаке, тем больший вклад этот признак вносит в ответ модели.
Важно! Признаки должны быть отмасштабированы!
Дополнительно можно использовать регулиризацию для отбора признаков.
Чем больше коэффициент при L1-регуляризаторе, тем меньше признаков будет использовать линейная модель (она занулит неинформативные признаки).

Обучим линейную модель и посмотрим на веса для признаков:
from sklearn.linear_model import LogisticRegression
from sklearn.preprocessing import StandardScaler
import seaborn as sns
scaler = StandardScaler()
x_train_scaled = scaler.fit_transform(x_train)
x_test_scaled = scaler.transform(x_test)
lr = LogisticRegression(max_iter=1000)
lr.fit(x_train_scaled, y_train)
df = pd.DataFrame({"name": x_train.columns, "coef": lr.coef_[0]}).sort_values(
"coef", key=abs, ascending=False
)
df["sign"] = ["neg" if x < 0 else "pos" for x in df["coef"]]
palette = {"neg": sns.xkcd_rgb["orange"], "pos": sns.xkcd_rgb["azure"]}
plt.figure(figsize=(8, 8))
sns.barplot(
data=df, y="name", x="coef", hue="sign", palette=palette, legend=False, orient="h"
)
plt.show()
Можно использовать SelectFromModel 🛠️[doc] из библиотеки Sklearn для отбора наиболее значимых признаков:
from sklearn.feature_selection import SelectFromModel
# selecting features based on importance weights
lr_selector = SelectFromModel(LogisticRegression(max_iter=1000))
lr_selector.fit(x_train_scaled, y_train)
x_train.columns[lr_selector.get_support()] # Get a mask of the features selected
Index(['Pclass', 'Sex', 'Age', 'SibSp', 'Title', 'sqrt_Fare', 'Cab_E'], dtype='object')
У случайного леса тоже есть свои важности признаков, только в этом случае важность признака определяется так: чем больше признак уменьшает impurity при разбиении, тем он более важный:
from sklearn.ensemble import RandomForestClassifier
rf = RandomForestClassifier(n_estimators=500, random_state=42)
rf.fit(x_train, y_train)
df = pd.DataFrame(
{"name": x_train.columns, "imp": rf.feature_importances_}
).sort_values("imp", key=abs, ascending=False)
df["sign"] = ["neg" if x < 0 else "pos" for x in df["imp"]]
palette = {"neg": sns.xkcd_rgb["orange"], "pos": sns.xkcd_rgb["azure"]}
plt.figure(figsize=(8, 8))
sns.barplot(
data=df, y="name", x="imp", hue="sign", palette=palette, legend=False, orient="h"
)
plt.show()
# select features with RFC
rf = RandomForestClassifier(n_estimators=500, random_state=42)
rf_selector = SelectFromModel(rf)
rf_selector.fit(x_train, y_train) # Fit it on the training data
x_train.columns[rf_selector.get_support()]
Index(['Pclass', 'Sex', 'Age', 'Title', 'sqrt_Fare', 'random_1', 'random_3',
'random_4'],
dtype='object')
Такие же важности можно вытащить из градиентного бустинга. Рассмотрим на примере CatBoost:
!pip install -q catboost
━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━ 98.2/98.2 MB 9.4 MB/s eta 0:00:00
from catboost import CatBoostClassifier
model = CatBoostClassifier(random_state=42, thread_count=-1)
model.fit(
x_train,
y_train,
eval_set=(x_test, y_test),
verbose=100,
plot=False,
early_stopping_rounds=100,
)
Learning rate set to 0.029139 0: learn: 0.6801871 test: 0.6814320 best: 0.6814320 (0) total: 47.3ms remaining: 47.2s 100: learn: 0.3289676 test: 0.4295331 best: 0.4285715 (95) total: 175ms remaining: 1.56s 200: learn: 0.2694309 test: 0.4232609 best: 0.4215241 (180) total: 292ms remaining: 1.16s Stopped by overfitting detector (100 iterations wait) bestTest = 0.42152413 bestIteration = 180 Shrink model to first 181 iterations.
<catboost.core.CatBoostClassifier at 0x7b97f37267a0>
feature_importance = model.feature_importances_
sorted_idx = np.argsort(feature_importance)
fig = plt.figure(figsize=(10, 5))
plt.barh(range(len(sorted_idx)), feature_importance[sorted_idx], align="center")
plt.yticks(range(len(sorted_idx)), np.array(x.columns)[sorted_idx])
plt.title("Feature Importance catboost")
plt.show()
В CatBoost также есть свой метод отбора признаков select_features 🛠️[doc]. Можно указать, какие признаки рассматривать для отбора (мы рассмотрим все признаки, параметр features_for_select) и количество признаков, которые нужно отобрать — num_features_to_select. Для примера отберем 5 признаков:
summary = model.select_features(
x_train,
y_train,
eval_set=(x_test, y_test),
features_for_select="0-22",
num_features_to_select=5,
steps=1,
train_final_model=False,
logging_level="Silent",
)
print(summary["selected_features_names"])
['Pclass', 'Sex', 'Title', 'Cabin_Number', 'sqrt_Fare']
Для определения важности признака можно перемешать его значения. Это не изменит форму распределения признака, но сделает его бессмысленным. По качеству модели мы можем оценить, каков был вклад признака до изменения: если качество упало сильно, то он был значимым.
В Sklearn это реализовано как отдельный класс permutation_importance 🛠️[doc].
Изменение качества модели рассчитывается на объектах, которые не вошли в обучающую выборку, а остались для валидации (не путать с тестовой выборкой, которую мы не трогаем).
У данного метода есть три особенности:
permutation_importance имеет параметр n_repeats, который определяет, сколько раз признак будет перемешиваться случайным образом.Для борьбы с неадекватной оценкой коррелированных признаков можно собрать коррелированные признаки в кластеры и оставить только один признак для каждого кластера 🛠️[doc].
Одна из вариаций permutation importance подразумевает обучение модели заново после каждого перемешивания признаков. Это дает чуть более адекватную оценку важности, но очень долго.
Сделаем permutation importance:
Ниже выполняются эти же операции, но уже с линейной моделью:
from sklearn.inspection import permutation_importance
model = LogisticRegression()
model.fit(x_train_scaled, y_train)
r = permutation_importance(model, x_test_scaled, y_test, n_repeats=100, random_state=42)
df = pd.DataFrame({"name": x_train.columns, "imp": r.importances_mean}).sort_values(
"imp", ascending=False
)
df["sign"] = ["neg" if x < 0 else "pos" for x in df["imp"]]
palette = {"neg": sns.xkcd_rgb["orange"], "pos": sns.xkcd_rgb["azure"]}
plt.figure(figsize=(8, 8))
sns.barplot(
data=df, y="name", x="imp", hue="sign", palette=palette, legend=False, orient="h"
)
plt.show()
И со случайным лесом:
model = RandomForestClassifier(n_estimators=500, random_state=42)
model.fit(x_train, y_train)
r = permutation_importance(model, x_test, y_test, n_repeats=100, random_state=42)
df = pd.DataFrame({"name": x_train.columns, "imp": r.importances_mean}).sort_values(
"imp", ascending=False
)
df["sign"] = ["neg" if x < 0 else "pos" for x in df["imp"]]
palette = {"neg": sns.xkcd_rgb["orange"], "pos": sns.xkcd_rgb["azure"]}
plt.figure(figsize=(8, 8))
sns.barplot(
data=df, y="name", x="imp", hue="sign", palette=palette, legend=False, orient="h"
)
plt.show()
Как видно, у некоторых признаков при переходе от древесных моделей к линейным важность признаков изменилась
Boruta развивает идею Permutation.
Увеличим наш датасет, добавив в него "теневые признаки" $-$ перемешанные реальные. Таким образом, наш датасет точно будет содержать хорошие признаки (мы ничего не удаляем).
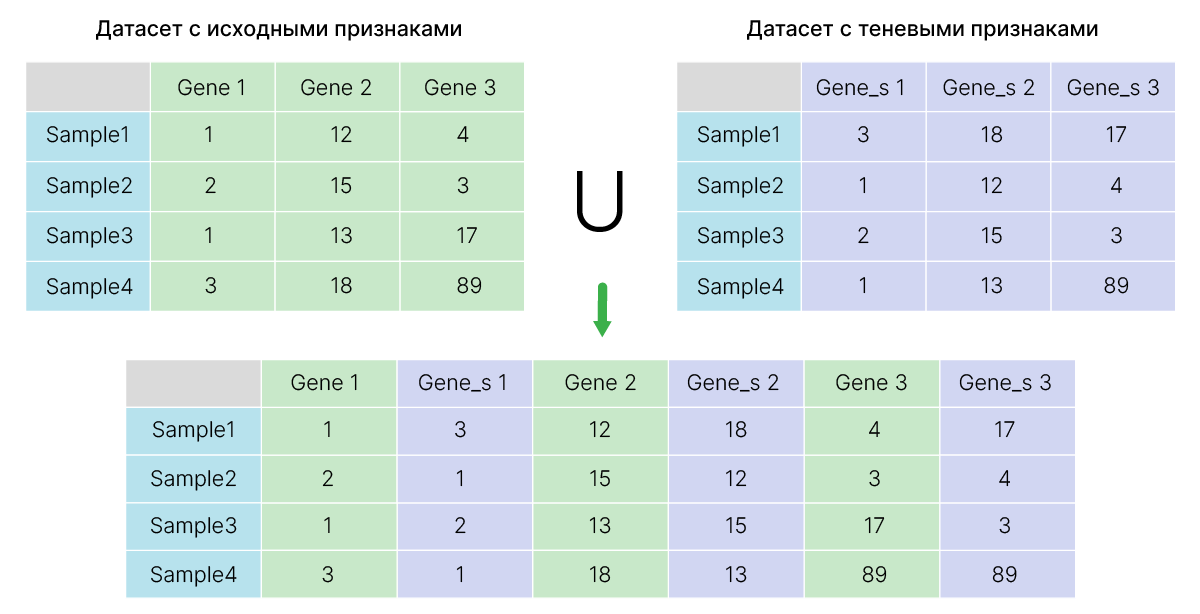
После этого обучаем модель и отбираем те признаки, чей feature_importance больше, чем у лучшего из теневых. Повторяем процедуру несколько раз для того, чтобы удалить случайные скачки качества.
Таким образом, для каждого признака мы будем знать, сколько раз мы его отобрали. Получаем распределение. Самая большая неопределенность будет в середине (вероятность отобрать = 0.5):
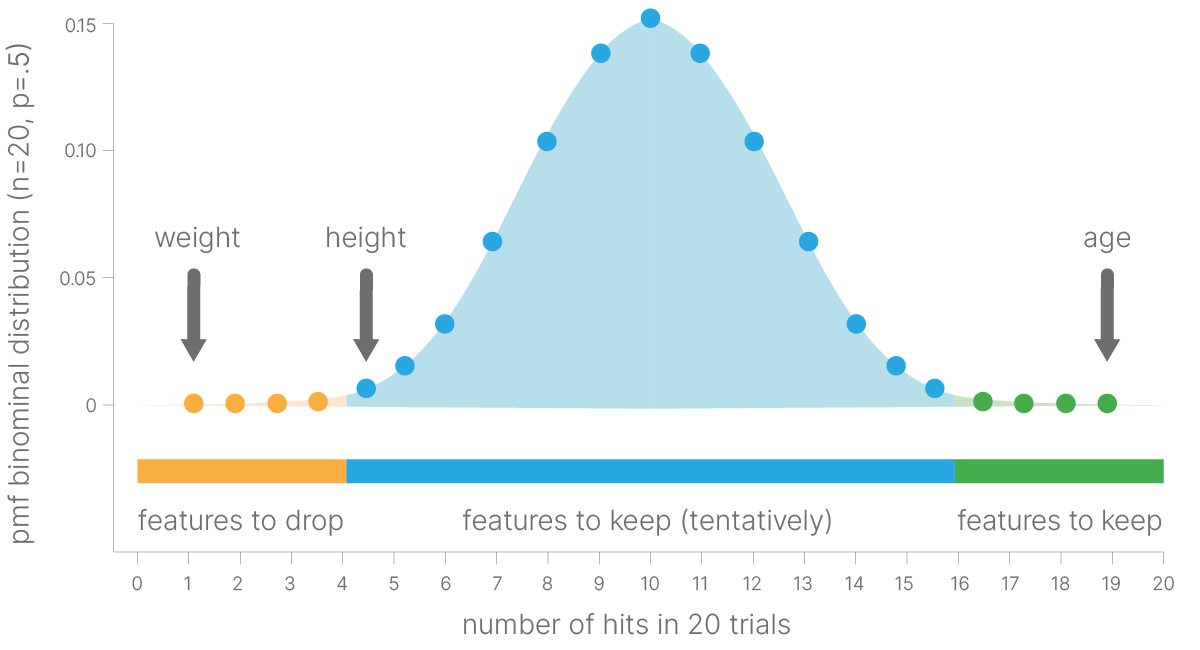
Набор (в нашем случае из 20) испытаний Бернулли — это биномиальное распределение. Со значимостью, допустим, 0.05 берем все из «хорошего хвоста» и отбрасываем из «плохого хвоста». С признаками из середины колокола ничего особо не сделаешь: увеличение числа итераций приведет к ужатию колокола, но глобально не поможет.
Если нам нужна хорошо интерпретируемая модель, то надо брать только точно хорошие признаки.
Будем использовать реализацию boruta_py 🐾[git]. Возьмем Random Forest модель.
model = RandomForestClassifier(n_estimators=500, random_state=42)
model.fit(x_train, y_train)
RandomForestClassifier(n_estimators=500, random_state=42)In a Jupyter environment, please rerun this cell to show the HTML representation or trust the notebook.
RandomForestClassifier(n_estimators=500, random_state=42)
Попробуем применить алгоритм Boruta к этому же датасету и сократить количество признаков. Оценим текущее качество модели, используя ROC-AUC:
pred = model.predict_proba(x_test)[:, 1]
r1 = roc_auc_score(y_score=pred, y_true=y_test)
print(f"ROC-AUC: {r1:.4f}")
ROC-AUC: 0.8868
Загрузим Boruta:
!pip install -q boruta
━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━━ 56.6/56.6 kB 1.1 MB/s eta 0:00:00
Питоновская реализация Boruta соответствует API sklearn и может использоваться как в конвейере, так и самостоятельно.
Boruta работает с numpy.array, поэтому используем .values() :
print(type(x_train))
print(type(x_train.values))
<class 'pandas.core.frame.DataFrame'> <class 'numpy.ndarray'>
Проведем оценку признаков при помощи Boruta.
from boruta import BorutaPy
# for NumPy and Boruta compatibility
np.int = np.int64
np.float = np.float64
np.bool = np.bool_
# define Boruta feature selection method
model = RandomForestClassifier(n_estimators=500, random_state=42)
feat_selector = BorutaPy(model, n_estimators=100, verbose=1, random_state=42)
# find all relevant features
feat_selector.fit(x_train.values, y_train.values)
Iteration: 1 / 100 Iteration: 2 / 100 Iteration: 3 / 100 Iteration: 4 / 100 Iteration: 5 / 100 Iteration: 6 / 100 Iteration: 7 / 100 Iteration: 8 / 100 Iteration: 9 / 100 Iteration: 10 / 100 Iteration: 11 / 100 Iteration: 12 / 100 Iteration: 13 / 100 Iteration: 14 / 100 Iteration: 15 / 100 Iteration: 16 / 100 Iteration: 17 / 100 Iteration: 18 / 100 Iteration: 19 / 100 Iteration: 20 / 100 Iteration: 21 / 100 Iteration: 22 / 100 Iteration: 23 / 100 Iteration: 24 / 100 Iteration: 25 / 100 Iteration: 26 / 100 BorutaPy finished running. Iteration: 27 / 100 Confirmed: 4 Tentative: 0 Rejected: 19
BorutaPy(estimator=RandomForestClassifier(random_state=RandomState(MT19937) at 0x7B97F29F9540),
n_estimators=100, random_state=RandomState(MT19937) at 0x7B97F29F9540,
verbose=1)In a Jupyter environment, please rerun this cell to show the HTML representation or trust the notebook. BorutaPy(estimator=RandomForestClassifier(random_state=RandomState(MT19937) at 0x7B97F29F9540),
n_estimators=100, random_state=RandomState(MT19937) at 0x7B97F29F9540,
verbose=1)RandomForestClassifier(random_state=RandomState(MT19937) at 0x7B97F29F9540)
RandomForestClassifier(random_state=RandomState(MT19937) at 0x7B97F29F9540)
Посмотрим, как оценила признаки Boruta:
keep = x_train.columns[feat_selector.support_].to_list()
normal = x_train.columns[feat_selector.support_weak_].to_list()
drop = x_train.columns[
~(feat_selector.support_ | feat_selector.support_weak_)
].to_list()
print("features keep:", keep)
print("features normal:", normal)
print("features drop:", drop)
features keep: ['Sex', 'Age', 'Title', 'sqrt_Fare'] features normal: [] features drop: ['Pclass', 'SibSp', 'Parch', 'Embarked', 'Cabin_Number', 'Cab_A', 'Cab_B', 'Cab_C', 'Cab_D', 'Cab_E', 'Cab_F', 'Cab_G', 'Cab_T', 'Cab_nan', 'random_1', 'random_2', 'random_3', 'random_4', 'random_5']
Посмотрим на более подробную информацию о результатах отбора:
feature_ranks = list(
zip(x_train.columns, feat_selector.ranking_, feat_selector.support_)
)
df = pd.DataFrame(feature_ranks, columns=["feature", "rank", "boruta_keep"])
df.sort_values("rank")
| feature | rank | boruta_keep | |
|---|---|---|---|
| 1 | Sex | 1 | True |
| 2 | Age | 1 | True |
| 6 | Title | 1 | True |
| 8 | sqrt_Fare | 1 | True |
| 0 | Pclass | 2 | False |
| 18 | random_1 | 3 | False |
| 7 | Cabin_Number | 4 | False |
| 20 | random_3 | 5 | False |
| 3 | SibSp | 6 | False |
| 21 | random_4 | 7 | False |
| 17 | Cab_nan | 8 | False |
| 22 | random_5 | 9 | False |
| 5 | Embarked | 10 | False |
| 4 | Parch | 11 | False |
| 19 | random_2 | 12 | False |
| 13 | Cab_E | 13 | False |
| 10 | Cab_B | 14 | False |
| 11 | Cab_C | 15 | False |
| 12 | Cab_D | 16 | False |
| 9 | Cab_A | 17 | False |
| 14 | Cab_F | 18 | False |
| 15 | Cab_G | 19 | False |
| 16 | Cab_T | 20 | False |
Boruta из 23 признаков предлагает оставить только 4: Sex, Age, Title, sqrt_Fare.
Зная информацию о ранге признака, мы можем взять больше признаков для обучения. Также обратите внимание, что случайный признак находится довольно высоко.
Обучим алгоритм на признаках, отобранных Boruta. Давайте проверим, можем ли мы добиться такого же результата, если проанализируем и уберем «лишние» данные?
# Build the model with the random forest algorithm
model = RandomForestClassifier(n_estimators=500, random_state=42)
model.fit(x_train[keep], y_train)
pred = model.predict_proba(x_test[keep])[:, 1]
r1 = roc_auc_score(y_score=pred, y_true=y_test)
print(f"ROC-AUC: {r1:.4f}")
ROC-AUC: 0.8702
Качество упало, но не так сильно. Можно поэкспериментировать с добавлением признаков более низкого ранга.
Жадные методы отбора признаков по сути являются надстройками над методами обучения моделей. Они перебирают различные подмножества признаков и выбирают то из них, которое дает наилучшее качество определённой модели машинного обучения.
Данный процесс устроен следующим образом:
Обучение модели считается черным ящиком, который на вход принимает информацию о том, какие из его признаков можно использовать при обучении модели, обучает модель, и дальше каким-то методом оценивается качество такой модели, например, по отложенной выборке или кросс-валидации. Таким образом, задача, которую необходимо решить — это оптимизация функционала качества модели по подмножеству признаков.
Полный перебор
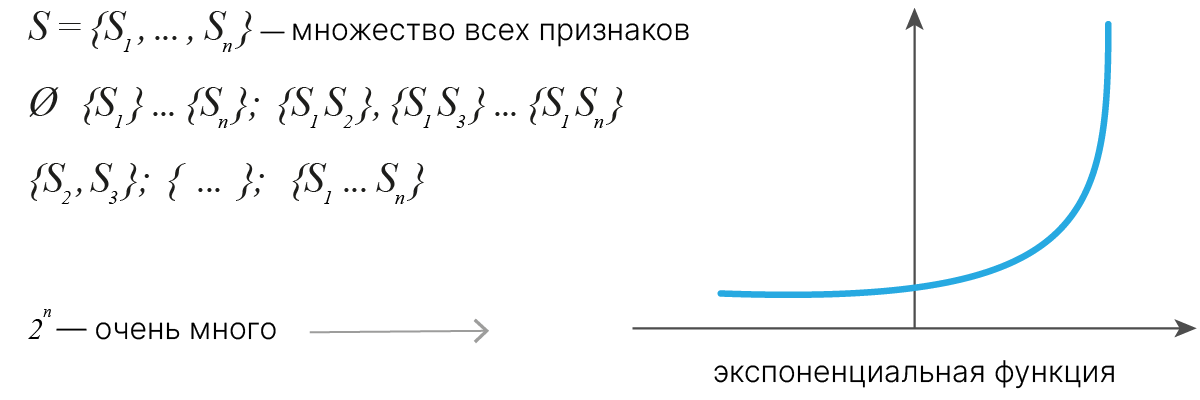
Самый простой способ решения данной задачи — это полный перебор всех подмножеств признаков и оценивание качества на каждом подмножестве. Итоговое подмножество — то, на котором качество модели наилучшее. Этот перебор можно структурировать и перебирать подмножества последовательно: сначала те, которые имеют мощность 1 (наборы из 1 признака), потом наборы мощности 2, и так далее. Это подход очень хороший, он найдет оптимальное подмножество признаков, но при этом очень сложный, поскольку всего таких подмножеств $2^d -1$, где $d$ — число признаков (пустое множество не рассматриваем). Если признаков много — сотни или тысячи, то такой перебор невозможен: он займет слишком много времени, возможно, сотни лет или больше. Поэтому, такой метод подходит либо при небольшом количестве признаков, либо если известно, что информативных признаков очень мало, единицы.
Жадное добавление
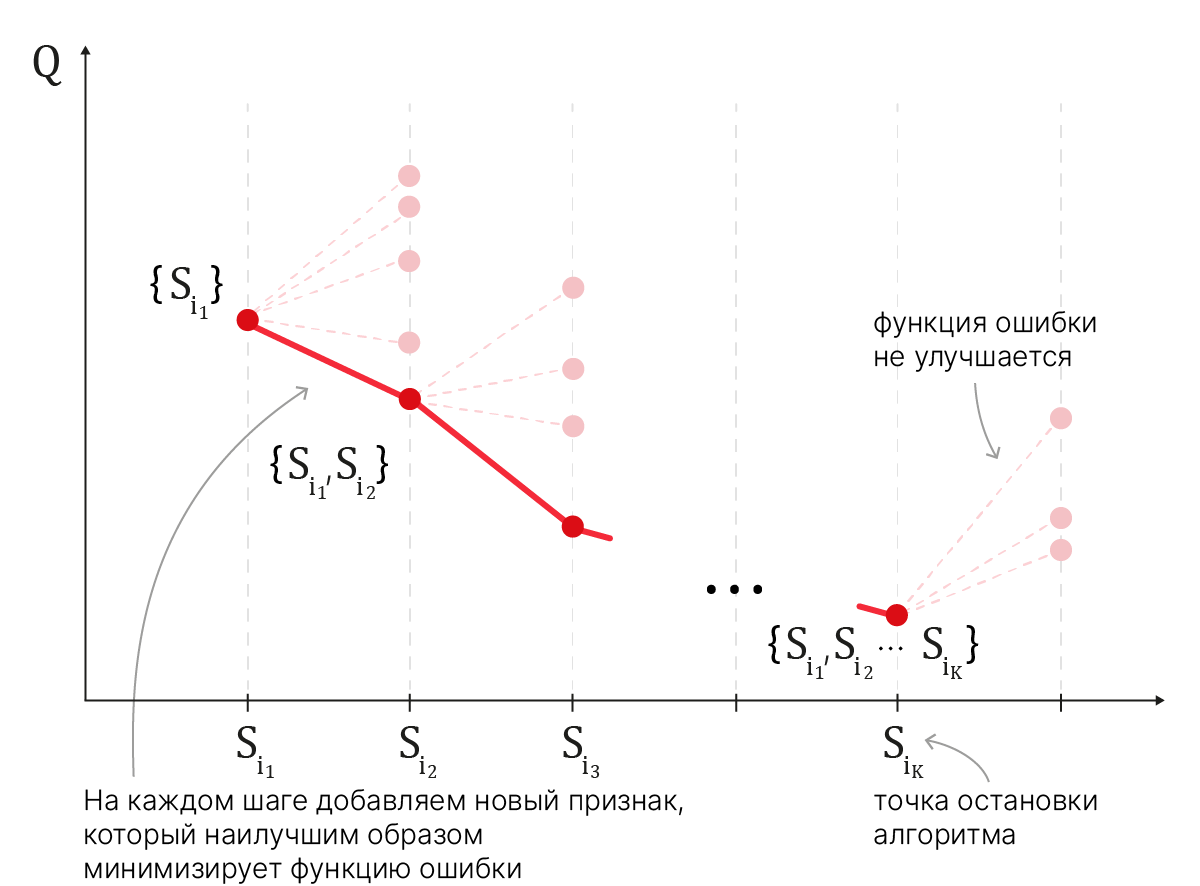
Жадная стратегия используется всегда, когда полный перебор не подходит для решения задачи. Например, может оказаться неплохой стратегия жадного наращивания (жадного добавления). Сначала находится один признак, который дает наилучшее качество модели (наименьшую ошибку $Q$):
$i_1 = \text{argmin} Q(i)$.
Тогда множество, состоящее из этого признака:
$J_1 = {i_1}$
Дальше к этому множеству добавляется еще один признак так, чтобы как можно сильнее уменьшить ошибку модели:
$i_2 = \text{argmin} Q(i_1,i)$, $J_2 ={i_1,i_2}$.
Далее каждый раз добавляется по одному признаку, образуются множества $J_3 , J_4 , . . . .$ Если в какой-то момент невозможно добавить новый признак так, чтобы уменьшить ошибку, процедура останавливается. Жадность процедуры заключается в том, что как только какой-то признак попадает в оптимальное множество, его нельзя оттуда удалить.
from mlxtend.feature_selection import SequentialFeatureSelector
from sklearn.ensemble import RandomForestClassifier
from sklearn.model_selection import KFold
sfs = SequentialFeatureSelector(
RandomForestClassifier(random_state=42),
k_features=8, # number of features to select
forward=True, # True - add, False - del
floating=False,
scoring="accuracy",
cv=KFold(n_splits=5, shuffle=True, random_state=42),
)
sfs.fit(x_train, y_train)
df = pd.DataFrame.from_dict(sfs.get_metric_dict()).T
df.head(df.shape[0])
| feature_idx | cv_scores | avg_score | feature_names | ci_bound | std_dev | std_err | |
|---|---|---|---|---|---|---|---|
| 1 | (1,) | [0.8181818181818182, 0.7482517482517482, 0.753... | 0.787935 | (Sex,) | 0.040774 | 0.031724 | 0.015862 |
| 2 | (1, 4) | [0.8181818181818182, 0.7552447552447552, 0.767... | 0.794967 | (Sex, Parch) | 0.037359 | 0.029067 | 0.014533 |
| 3 | (1, 4, 8) | [0.8041958041958042, 0.8181818181818182, 0.774... | 0.814616 | (Sex, Parch, sqrt_Fare) | 0.032575 | 0.025345 | 0.012672 |
| 4 | (1, 4, 8, 14) | [0.7972027972027972, 0.8251748251748252, 0.781... | 0.82025 | (Sex, Parch, sqrt_Fare, Cab_F) | 0.037555 | 0.029219 | 0.014609 |
| 5 | (1, 4, 8, 14, 16) | [0.7972027972027972, 0.8321678321678322, 0.781... | 0.82024 | (Sex, Parch, sqrt_Fare, Cab_F, Cab_T) | 0.037626 | 0.029274 | 0.014637 |
| 6 | (0, 1, 4, 8, 14, 16) | [0.8181818181818182, 0.8251748251748252, 0.781... | 0.823038 | (Pclass, Sex, Parch, sqrt_Fare, Cab_F, Cab_T) | 0.032031 | 0.024921 | 0.012461 |
| 7 | (0, 1, 4, 8, 14, 15, 16) | [0.8111888111888111, 0.8251748251748252, 0.781... | 0.825864 | (Pclass, Sex, Parch, sqrt_Fare, Cab_F, Cab_G, ... | 0.038813 | 0.030198 | 0.015099 |
| 8 | (0, 1, 4, 8, 13, 14, 15, 16) | [0.8111888111888111, 0.8251748251748252, 0.767... | 0.824456 | (Pclass, Sex, Parch, sqrt_Fare, Cab_E, Cab_F, ... | 0.045308 | 0.035251 | 0.017626 |
df["feature_names"].values[-1]
('Pclass', 'Sex', 'Parch', 'sqrt_Fare', 'Cab_E', 'Cab_F', 'Cab_G', 'Cab_T')
import matplotlib.pyplot as plt
from mlxtend.plotting import plot_sequential_feature_selection as plot_sfs
plot_sfs(sfs.get_metric_dict(), kind="std_dev")
plt.title("Sequential Forward Selection (StdDev)")
plt.grid()
plt.show()
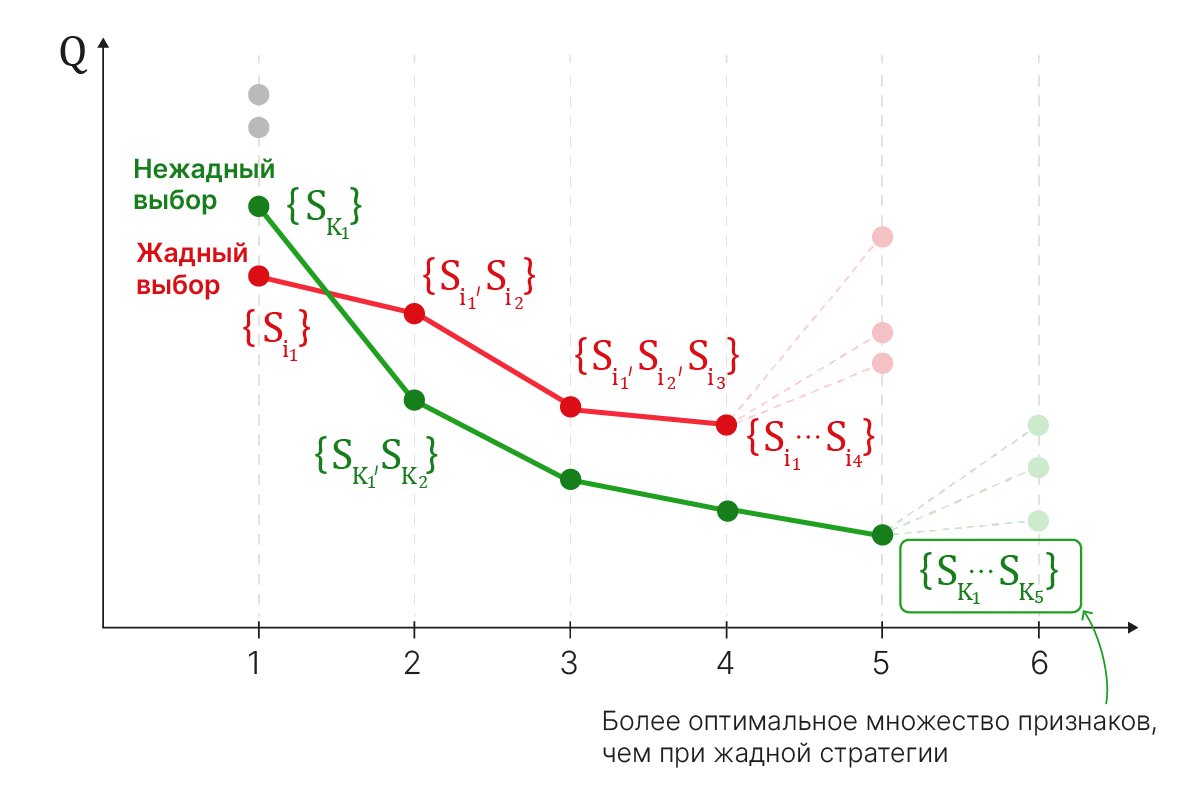
Описанный выше подход довольно быстрый: в нем столько итераций, сколько признаков в выборке. Но при этом он слишком жадный: перебирается слишком мало вариантов, и мы можем оказаться в плохой локальной точке. Процедуру можно усложнить. Один из подходов к усложнению — это алгоритм ADD-DEL, который не только добавляет, но и удаляет признаки из оптимального множества.
Алгоритм начинается с процедуры жадного добавления. Множество признаков наращивается до тех пор, пока получается уменьшить ошибку, затем признаки жадно удаляются из подмножества, то есть перебираются все возможные варианты удаления признака, оценивается ошибка и удаляется тот признак, удаление которого приводит к наибольшему уменьшению ошибки на выборке. Эта процедура повторяет добавление и удаление признаков до тех пор, пока уменьшается ошибка. Алгоритм ADD-DEL всё еще жадный, но при этом он менее жадный, чем предыдущий, поскольку может исправлять ошибки, сделанные в начале перебора: если вначале был добавлен неинформативный признак, то на этапе удаления от него можно избавиться.
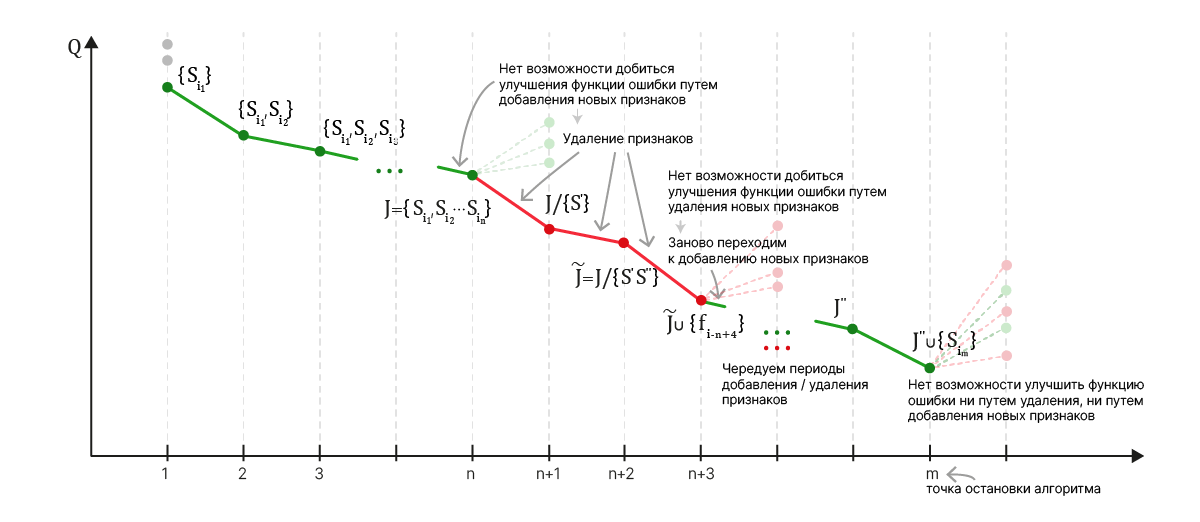
sffs = SequentialFeatureSelector(
RandomForestClassifier(random_state=42), # represents the classifier
k_features=8, # the number of features you want to select
forward=True, # add features
floating=True, # remove features
scoring="accuracy", # means that the selection will be decided by the accuracy of the classifier.
cv=KFold(n_splits=5, shuffle=True, random_state=42),
)
sffs.fit(x_train.values, y_train) # performs the actual SFFS algorithm
df = pd.DataFrame.from_dict(sffs.get_metric_dict()).T
df.head(df.shape[0])
| feature_idx | cv_scores | avg_score | feature_names | ci_bound | std_dev | std_err | |
|---|---|---|---|---|---|---|---|
| 1 | (1,) | [0.8181818181818182, 0.7482517482517482, 0.753... | 0.787935 | (1,) | 0.040774 | 0.031724 | 0.015862 |
| 2 | (1, 4) | [0.8181818181818182, 0.7552447552447552, 0.767... | 0.794967 | (1, 4) | 0.037359 | 0.029067 | 0.014533 |
| 3 | (1, 4, 8) | [0.8041958041958042, 0.8181818181818182, 0.774... | 0.814616 | (1, 4, 8) | 0.032575 | 0.025345 | 0.012672 |
| 4 | (1, 4, 8, 14) | [0.7972027972027972, 0.8251748251748252, 0.781... | 0.82025 | (1, 4, 8, 14) | 0.037555 | 0.029219 | 0.014609 |
| 5 | (1, 4, 8, 14, 16) | [0.7972027972027972, 0.8321678321678322, 0.781... | 0.82024 | (1, 4, 8, 14, 16) | 0.037626 | 0.029274 | 0.014637 |
| 6 | (0, 1, 4, 8, 14, 16) | [0.8181818181818182, 0.8251748251748252, 0.781... | 0.823038 | (0, 1, 4, 8, 14, 16) | 0.032031 | 0.024921 | 0.012461 |
| 7 | (0, 1, 4, 8, 14, 15, 16) | [0.8111888111888111, 0.8251748251748252, 0.781... | 0.825864 | (0, 1, 4, 8, 14, 15, 16) | 0.038813 | 0.030198 | 0.015099 |
| 8 | (0, 1, 4, 8, 13, 14, 15, 16) | [0.8111888111888111, 0.8251748251748252, 0.767... | 0.824456 | (0, 1, 4, 8, 13, 14, 15, 16) | 0.045308 | 0.035251 | 0.017626 |
x.iloc[:, list(df["feature_idx"].values[-1])].columns
Index(['Pclass', 'Sex', 'Parch', 'sqrt_Fare', 'Cab_E', 'Cab_F', 'Cab_G',
'Cab_T'],
dtype='object')
plot_sfs(sffs.get_metric_dict(), kind="std_dev")
plt.title("Sequential Forward Selection (StdDev)")
plt.grid()
plt.show()
Важно помнить, что нельзя отбирать признаки на том же наборе данных, на котором тестируетесь.
Иначе получите завышенное качество вашей модели.
Часто мы хотим данные из пространства высокой размерности перевести в пространство более низкой размености с сохранением одного или нескольких свойств, например:
Мотивация понижения размерности:
Многие алгоритмы показывают себя плохо на пространствах большой размерности (проклятие размерности 📚[wiki]).
Некоторые просто будут значительно дольше работать, при этом качество их работы не изменится от уменьшения размерности.
Понижение размерности позволяет использовать память более эффективно и подавать модели на обучение за один раз больше объектов.
Избавиться от шумовых признаков.
Задача визуализации: хочется взглянуть на наши объекты, а делать это в 100-мерном или 100000-мерном пространстве невозможно.
Удаление выбросов — в пространствах меньшей размерности можем их увидеть глазами.
Можем увидеть закономерности в данных: что они разделяются на явные кластеры и т. д.
В машинном обучении часто используют предположение о многообразии (manifold assumption). Это предположение о том, что между признаками, описывающими реальные объекты, существуют некоторые нетривиальные связи. Вследствие этого данные заполняют не весь объем пространства признаков, а лежат на некоторой поверхности — на многообразии (manifold).
Если предположение верно, то каждый объект может быть достаточно точно описан новыми признаками в пространстве значительно меньшей размерности, чем исходное пространство признаков.
При этом мы будем терять часть информации об объектах. Но при выполнении предположения о многообразии (а оно почти всегда выполняется) и при правильных настройках алгоритма понижения размерности эти потери будут незначительны.
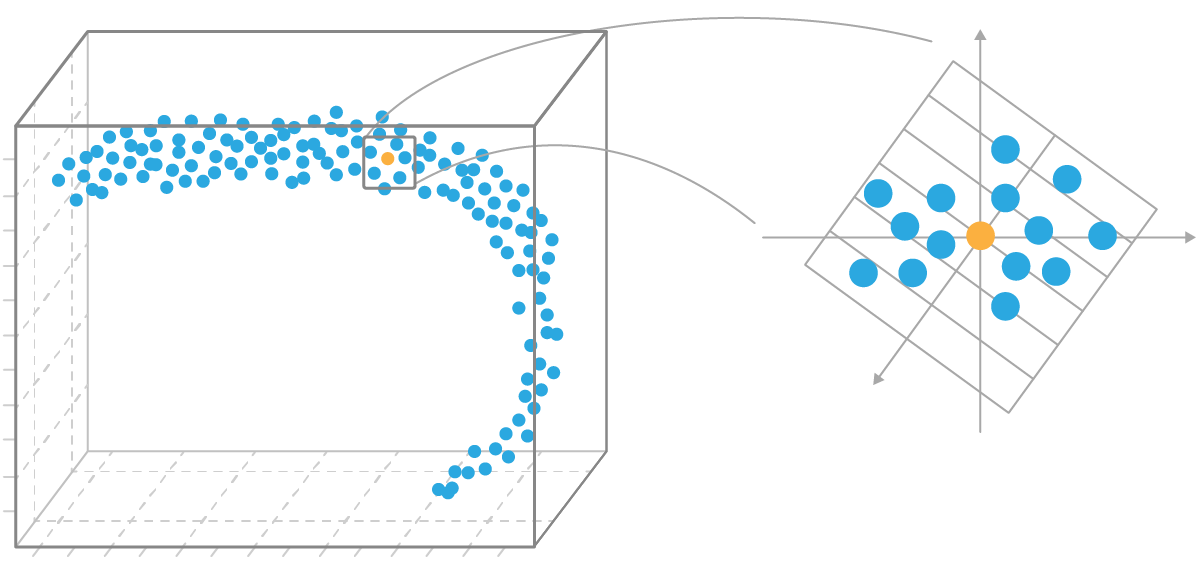
В большинстве случаев это действительно правда. Например, лица людей даже на фотографиях $300\times300$ лежат в пространстве меньшей размерности, нежели $90000$. Не каждая матрица размером $300\times300$, заполненная какими-то значениями от $0$ до $1$, даст нам изображение лица человека.
Метод главных компонент (Principal Component Analysys, PCA) — простейший линейный метод снижения размерности, описан Пирсоном в 1901 году 📚[book].
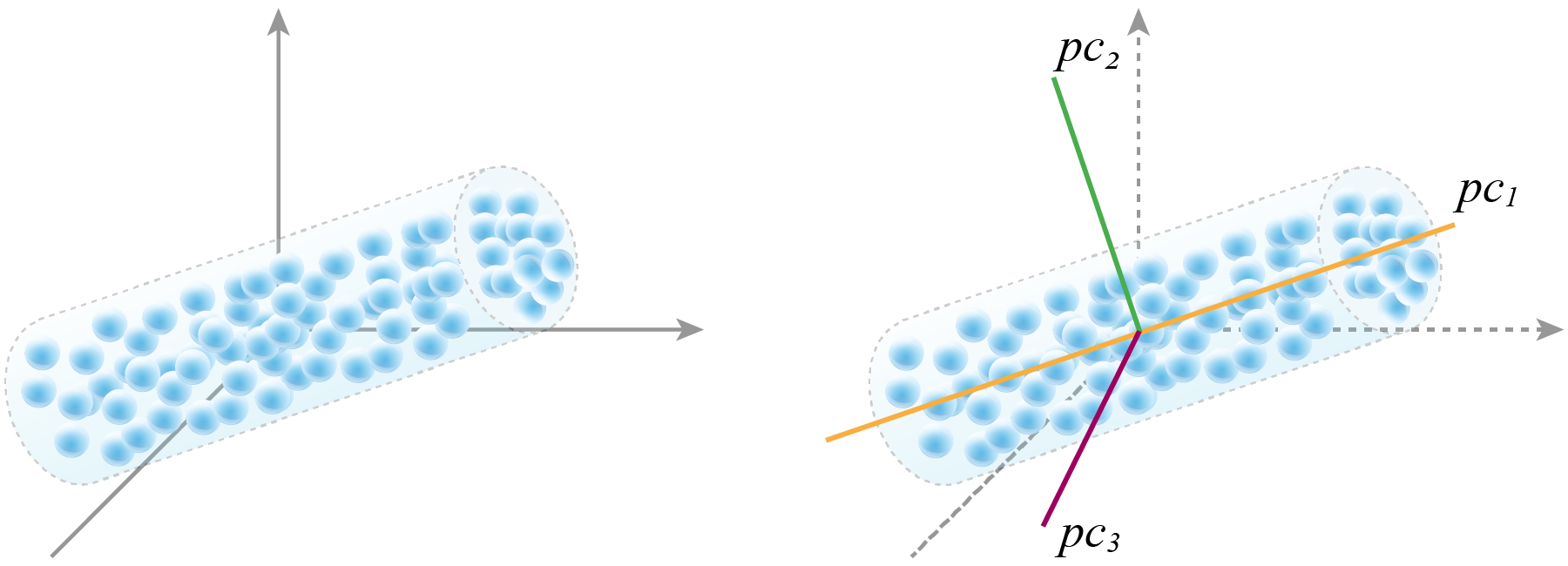
Рассмотрим простой пример для понимания интуиции, которая стоит за методом главных компонент.
У нас есть выборка объектов с двумя признаками $x_1$ и $x_2$, отобразим их на плоскости. Мы хотим, чтобы у нас был только один признак, который характеризует наши данные. Тогда наша задача — провести прямую через наши точки так, чтобы она наилучшим образом характеризовала наши данные
Проведем прямую вдоль максимального изменения данных и спроецируем точки на нее. Это и будет наша первая главная компонента.
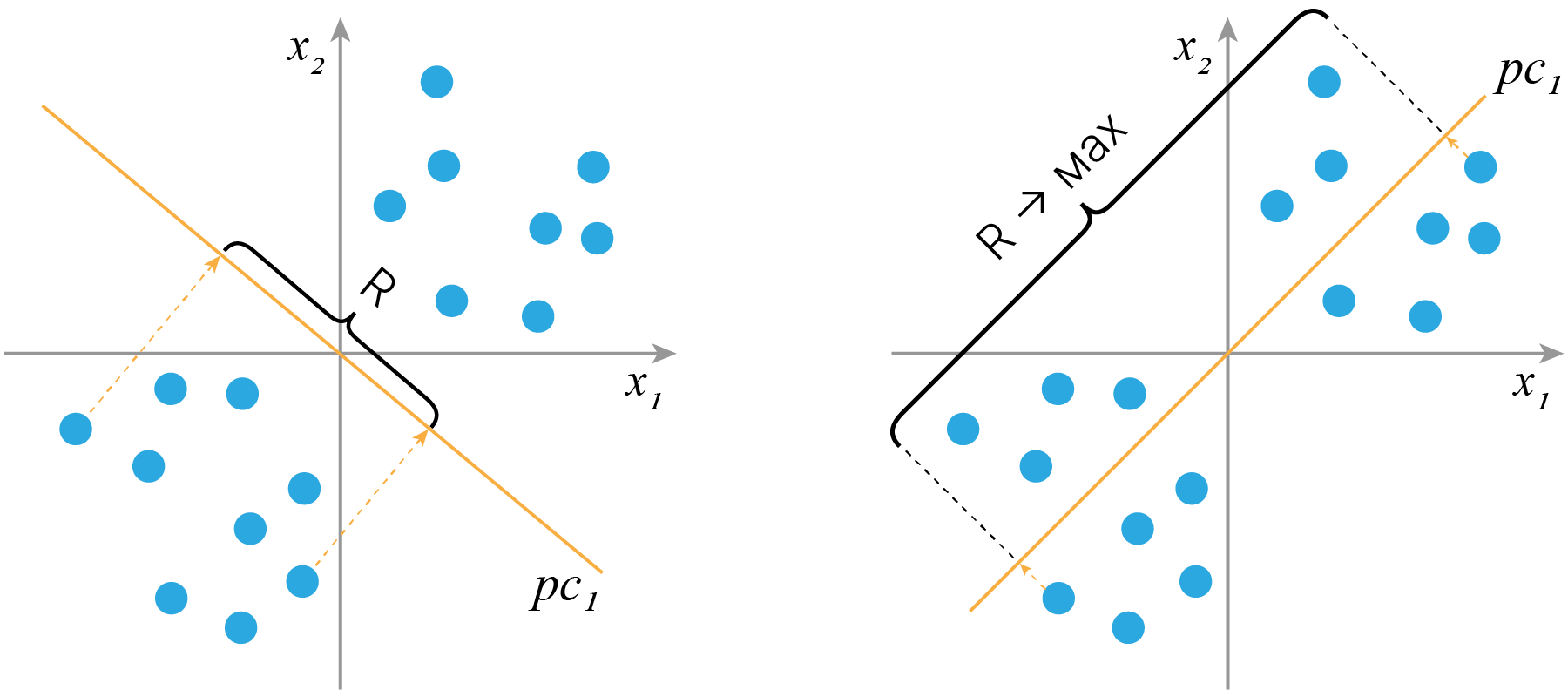
Теперь мы можем использовать только один признак, который лежит на новой оси, а любые отклонения от нее — это просто шум (в действительности, определить, что является шумом в данных — это нетривиальная задача).
Это то, как можно понимать интуицию работы PCA.
В более многомерном случае (например, при количестве признаков 200) мы бы делали то же самое:
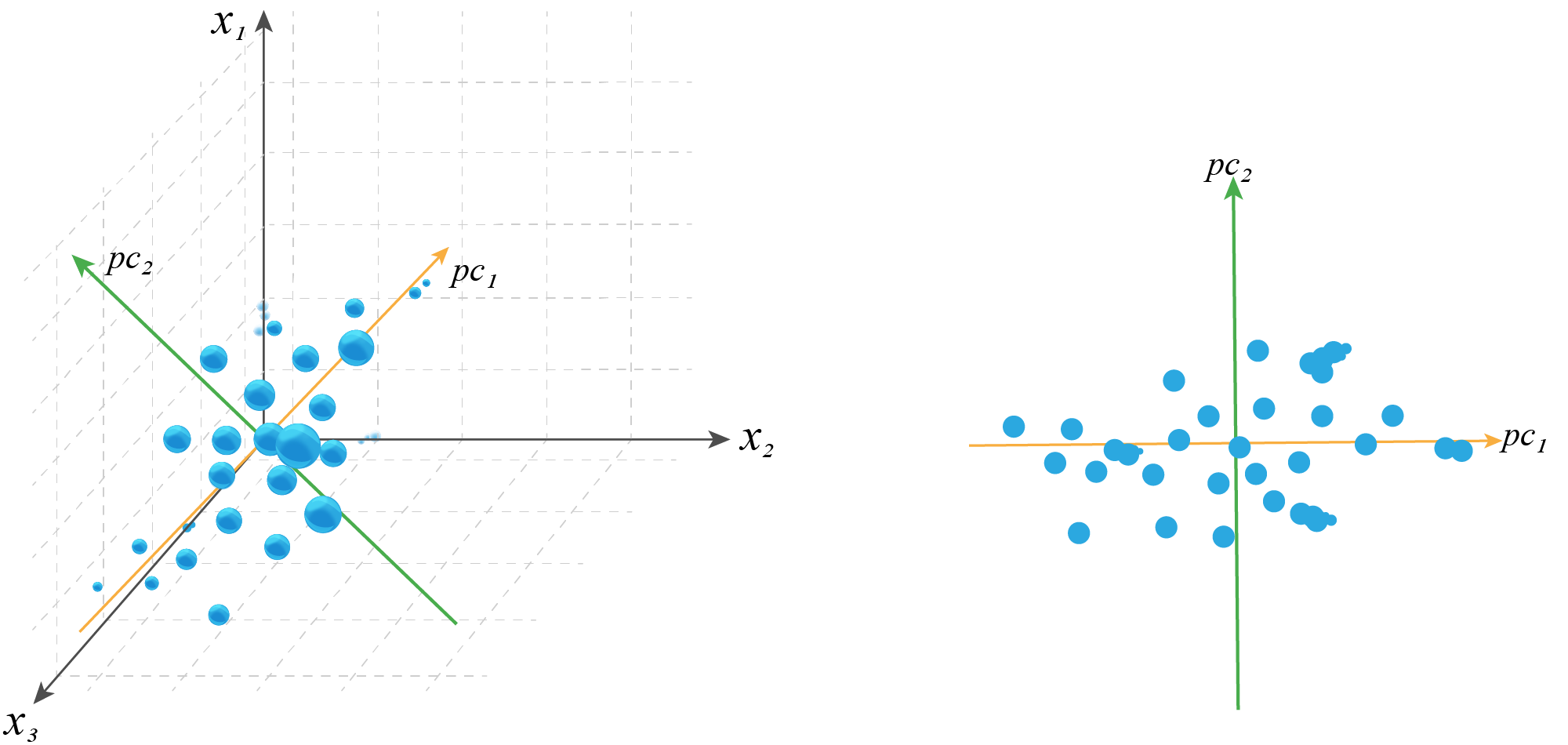
Чтобы понять, как использовать PCA на практике, найдем главные компоненты для датасета и посмотрим, как распределится между ними дисперсия.
Скачаем датасет, на котором польза от понижения размерности видна более явно.
В данном датасете 977 признаков (нам сейчас не важно, какие), характеризующие клетки крови.
Загрузим датасет и подготовим данные:
import numpy as np
import pandas as pd
from sklearn.model_selection import train_test_split
scRNAseq = pd.read_csv(
"https://edunet.kea.su/repo/EduNet-web_dependencies/datasets/scRNAseq_CITEseq.txt",
sep="\t",
)
x = scRNAseq.iloc[:, 0:-1] # features
y = scRNAseq.iloc[:, -1] # labels
# 2. taking the log of the data.
x = np.log(x + 1)
x_train, x_test, y_train, y_test = train_test_split(
x, y, test_size=0.2, random_state=42
)
print(f"dataset shape: {x.shape}")
x
dataset shape: (8617, 976)
| ABHD17A | ABHD5 | ABI3 | AC013264.2 | AC017104.6 | AC092580.4 | AC093673.5 | AC132872.2 | ACRBP | ACSL1 | ... | MOUSE_Rtn4 | MOUSE_Ppp1ca | MOUSE_Cald1 | MOUSE_Rps26 | MOUSE_Ctsl | MOUSE_Tnfrsf12a | MOUSE_Esd | MOUSE_Acta2 | MOUSE_Arpc2 | MOUSE_Calr | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| CTGTTTACACCGCTAG | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.000000 | 0.0 | ... | 4.406719 | 3.663562 | 5.087596 | 3.135494 | 4.897840 | 4.060443 | 4.077537 | 1.791759 | 4.204693 | 3.091042 |
| CTCTACGGTGTGGCTC | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.000000 | 0.0 | ... | 4.330733 | 4.219508 | 4.454347 | 4.110874 | 4.356709 | 4.025352 | 4.262680 | 4.248495 | 3.970292 | 3.526361 |
| AGCAGCCAGGCTCATT | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.693147 | 0.0 | ... | 4.634729 | 4.248495 | 4.477337 | 4.110874 | 4.727388 | 4.317488 | 3.637586 | 3.332205 | 4.234107 | 4.343805 |
| GAATAAGAGATCCCAT | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.000000 | 0.0 | ... | 4.605170 | 4.276666 | 4.477337 | 4.442651 | 4.219508 | 3.806662 | 4.394449 | 3.465736 | 4.143135 | 4.682131 |
| GTGCATAGTCATGCAT | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.000000 | 0.0 | ... | 4.276666 | 4.488636 | 4.859812 | 4.077537 | 3.178054 | 3.401197 | 2.639057 | 6.306275 | 4.543295 | 3.465736 |
| ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... |
| TTGCCGTGTAGATTAG | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.000000 | 0.0 | ... | 0.000000 | 0.000000 | 0.000000 | 0.000000 | 0.000000 | 0.000000 | 0.000000 | 0.693147 | 0.000000 | 0.000000 |
| GGCGTGTAGTGTACTC | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.000000 | 0.0 | ... | 0.000000 | 0.000000 | 0.000000 | 0.000000 | 0.693147 | 0.000000 | 0.000000 | 0.000000 | 0.000000 | 0.000000 |
| CGTATGCCGTCTTCTG | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.000000 | 0.0 | ... | 0.000000 | 0.000000 | 0.693147 | 0.000000 | 0.000000 | 0.000000 | 0.000000 | 0.000000 | 0.000000 | 0.693147 |
| TACACGACGCTCTTCC | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.000000 | 0.0 | ... | 0.693147 | 0.693147 | 0.000000 | 0.000000 | 1.098612 | 0.693147 | 0.693147 | 1.098612 | 1.098612 | 0.000000 |
| ACACGACGCTCTTCCG | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.000000 | 0.0 | ... | 0.000000 | 0.000000 | 0.693147 | 0.000000 | 0.000000 | 0.000000 | 0.000000 | 1.098612 | 0.000000 | 0.693147 |
8617 rows × 976 columns
Полезной информацией является коэффициент объясненной дисперсии (explained variance ratio) главной компоненты. Этот коэффициент является отношением между дисперсией главной компоненты и суммой дисперсий всех главных компонент. Он указывает долю выборочной дисперсии, которая лежит вдоль оси каждой главной компоненты.
В модуле PCA после fit можно получить explained variance ratio посредством обращения к полю explained_variance_ratio_, а explained variance — посредством обращения к полю explained_variance_.
Построим график с explained_variance_ratio_ для 10 компонент.
import sklearn
import matplotlib.pyplot as plt
from sklearn.decomposition import PCA
# performing PCA with default number of principal components.
pca = PCA(n_components=10)
pca.fit(x_train) # fitting our PCA model with the training data.
# calculating the explained variance of each of the components.
evr = pca.explained_variance_ratio_
# We are plotting the explained variance ratios.
plt.bar(range(1, evr.shape[0] + 1), evr)
plt.title("Variance by components")
plt.xlabel("Components")
plt.ylabel("Variance")
plt.show()
pca.explained_variance_.shape
(10,)
Из графика не совсем понятно, сколько компонент брать, резко доля объясняемой дисперсии меняется в районе 2-й компоненты. Посмотрим, сколько компонент нужно модели.
from sklearn.linear_model import LogisticRegression
pca = PCA(n_components=10)
pca.fit(x_train)
x_train_reduced = pca.transform(x_train)
x_test_reduced = pca.transform(x_test)
for i in range(1, 11):
model = LogisticRegression(max_iter=1000)
model.fit(x_train_reduced[:, :i], y_train)
print("%i first components %.2f" % (i, model.score(x_test_reduced[:, :i], y_test)))
1 first components 0.46 2 first components 0.78 3 first components 0.89 4 first components 0.93 5 first components 0.94 6 first components 0.94 7 first components 0.95 8 first components 0.96 9 first components 0.96 10 first components 0.96
Видим, что уже первых 8 компонент достаточно для достижения качества, которое далее не меняется.
Дело в том, что мы забыли сделать стандартизацию данных.
В датасете переменные имеют совершенно разные масштабы. Из-за этого часть из них "перетегягивает" на себя всю дисперсию.
В результате по доле дисперсии судить о важности компонент нельзя.
Сделаем предварительно стандартизацию:
# First, we import the StandardScaler module.
from sklearn.preprocessing import StandardScaler
# Next, we create a StandardScaler object called scaler by calling the
# StandardScaler() function.
scaler = StandardScaler()
# We then fit the scaling model to our training data.
x_train = scaler.fit_transform(x_train)
# We transform your test set by applying the same scaling model.
x_test = scaler.transform(x_test)
# performing PCA with default number of principal components.
pca = PCA(n_components=10)
pca.fit(x_train) # fitting our PCA model with the training data.
PCA(n_components=10)In a Jupyter environment, please rerun this cell to show the HTML representation or trust the notebook.
PCA(n_components=10)
В PCA.components_ лежат векторы главных компонент, расположенные построчно (n_components, n_features).
pca.components_.shape
(10, 976)
Обратите внимание, что PCA упорядочивает собственные векторы. Это значит, что собственный вектор, соответствующий главной компоненте и, соответственно, имеющей максимальную дисперсию, будет находиться в первой строке.
# calculating the explained variance of each of the components.
evr = pca.explained_variance_ratio_
# We are plotting the explained variance ratios.
plt.bar(range(evr.shape[0]), evr)
plt.title("Variance by components")
plt.xlabel("Components")
plt.ylabel("Variance")
plt.show()
Теперь видим, что не только первые две компоненты дают вклад. Это происходит потому, что переменные отмасштабированы.
Но и значимого спада в доле объясняемой дисперсии мы не видим, поэтому встает вопрос: как выбрать число компонент так, чтобы взять нужное и отсечь ненужное?
Подбирать число компонент можно по-разному
Мы можем выбрать порог по доле объясняемой дисперсии, например 95%. Это быстрый способ, но не ясно почему именно 95%, а не 90%.
pca = PCA(0.95)
pca.fit(x_train)
total_explained = np.cumsum(pca.explained_variance_ratio_)
plt.figure(figsize=(6, 6))
plt.plot(np.arange(1, len(total_explained) + 1), total_explained)
plt.axhline(y=0.95, color="red", linestyle="--")
chosen_number = np.argmax(total_explained >= 0.95) + 1
plt.axvline(x=chosen_number, color="red", linestyle="--")
plt.ylabel("Total Proportion of Explained Variance")
plt.xlabel("Number of Components")
print(f"{chosen_number} components to 95% explained_variance_ratio")
plt.show()
724 components to 95% explained_variance_ratio
Получилось 724 компоненты. При выборе другого порога получили бы другое количество компонент.
Можно построить график, отражающий, сколько дисперсии объясняет каждая из компонент, и на основе графика выбрать нужное число компонент.
Идея подхода простая: переход от компонент, объясняющих что-то важное в данных, к компонентам, объясняющим шум, должен сопровождаться резким снижением доли объясняемой дисперсии.
Или можно сказать иначе: выбранные нами компоненты должны быть устойчивы к добавлению шума в данные. Если мы нашли резкий скачок в доле объясняемой дисперсии, то маловероятно, что добавление шума этот скачок нивелирует — наш способ отбора компонент устойчив к шуму.
В идеальном случае график будет выглядеть как-то так. Но на практике таких склонов может не быть, а может быть несколько.
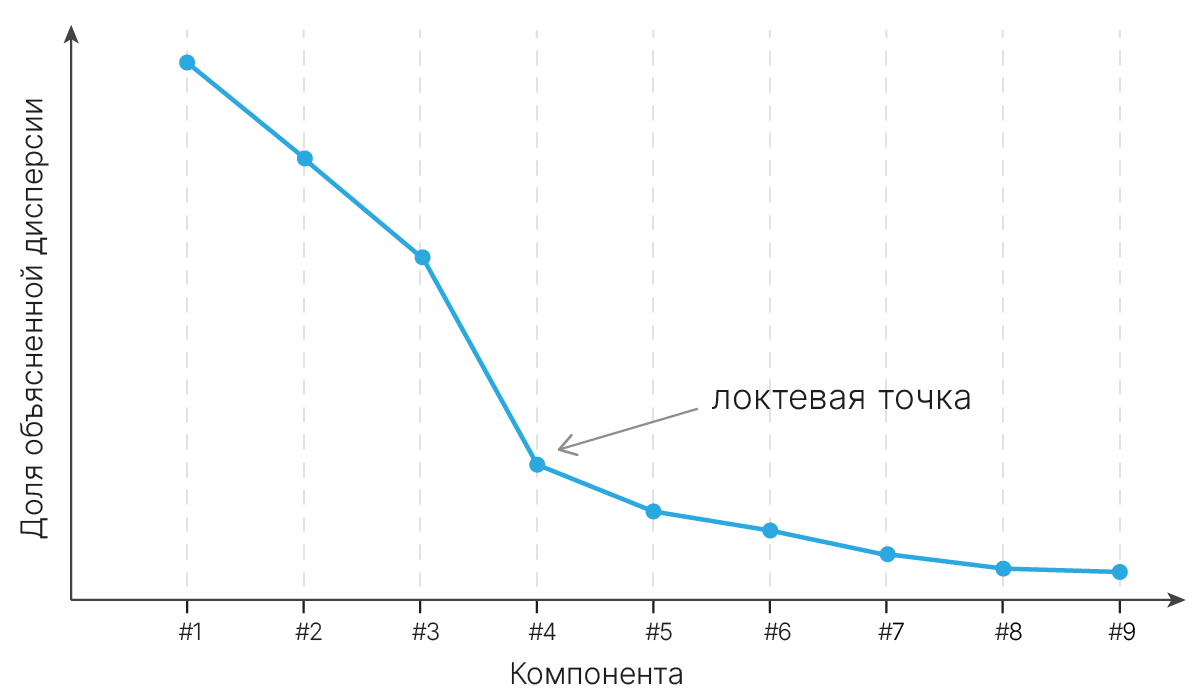
n_comp = 20
pca = PCA(n_comp)
pca.fit(x_train)
explained = pca.explained_variance_ratio_
plt.figure(figsize=(8, 6))
plt.scatter(np.arange(1, n_comp + 1), explained)
plt.plot(np.arange(1, n_comp + 1), explained)
plt.title("Dependence of variance on the number of components", size=14)
plt.xlabel("Num of components", size=14)
plt.ylabel("proportion of the explained variance", size=14)
plt.xticks(np.arange(1, n_comp + 1))
plt.show()
Из такого графика сложно понять, какое число компонент стоит выбрать.
Еще одним способом выбрать число главных компонент является перестановочный метод. Он заключается в следующем:
from tqdm import tqdm
def shuffle_dataset(dataset):
random_data = {
col: np.random.permutation(dataset[col].values) for col in dataset.columns
}
return pd.DataFrame(random_data)
def get_variance_by_chance(dataset, n_replics, n_components):
variance_explained_by_chance = np.empty((n_replics, n_components))
for i in tqdm(range(n_replics)):
random_data = shuffle_dataset(dataset)
random_pca = PCA(n_components=n_components)
variance_explained_by_chance[i, :] = random_pca.fit(
random_data
).explained_variance_ratio_
return variance_explained_by_chance
def calc_permutat_pval(real_values, permut_values, eps=None):
eps = eps or (1 / (permut_values.shape[0] * 10))
p_values = np.mean(permut_values >= real_values, axis=0) + eps
return p_values
def pca_analysis(dataset, title, n_components=None, n_replics=100):
n_components = n_components or dataset.shape[1]
explained_variance = (
PCA(n_components=n_components).fit(dataset).explained_variance_ratio_
)
variance_by_chance = get_variance_by_chance(dataset, n_replics, n_components)
p_values = calc_permutat_pval(explained_variance, variance_by_chance)
fig, (ax1, ax2) = plt.subplots(1, 2, figsize=(12, 6))
ax1.set_title(f"PCA {title}", size=15)
ax1.set_xlabel("Component number", size=15)
ax1.set_ylabel("Explained variance ratio", size=15)
ax1.plot(explained_variance, label="real", lw=3)
ax1.fill_between(
range(explained_variance.shape[0]),
np.mean(variance_by_chance, axis=0) + np.std(variance_by_chance, axis=0),
np.mean(variance_by_chance, axis=0) - np.std(variance_by_chance, axis=0),
color="red",
lw=3,
label="Chance",
)
ax2.set_title(f"PC significance, {title}", size=15)
ax2.set_xlabel("Component number", size=15)
ax2.set_ylabel("-log(pvalue + eps)", size=15)
p_values = -np.log10(p_values)
ax2.plot(p_values, lw=3)
ax2.hlines(
y=-np.log10(0.05),
xmin=0,
xmax=p_values.shape[0],
color="red",
linestyles="dashed",
lw=3,
)
fig.tight_layout()
return fig, (ax1, ax2)
np.random.seed(42)
pca_analysis(pd.DataFrame(x_train), "scRNAseq", n_replics=10, n_components=40)
plt.show()
100%|██████████| 10/10 [00:14<00:00, 1.44s/it]
Явно видим, что, согласно этому подходу, надо взять 30 компонент.
[arxiv] 🎓 Permutation methods for factor analysis and PCA — статья с рассуждениями на тему "Почему это работает?"
Загрузим данные биологического анализа пациентов с раком и без него.
В данном случае наши данные уже предварительно обработаны, поэтому стандартизацию проводить не будем
rnadataset = pd.read_table(
"https://edunet.kea.su/repo/EduNet-web_dependencies/datasets/rnaseq_data.tab.txt",
index_col=0,
header=None,
)
print("dataset shape: ", rnadataset.shape)
dataset shape: (358, 71586)
rnadataset.columns = list(rnadataset.columns[:-2]) + ["dataset", "sample type"]
rnadataset.head()
| 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | ... | 71577 | 71578 | 71579 | 71580 | 71581 | 71582 | 71583 | 71584 | dataset | sample type | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | |||||||||||||||||||||
| GSM1296956 | 13.374975 | 3.536581 | 13.644486 | 3.929925 | 5.485977 | 9.363128 | 13.134106 | 4.318162 | 10.050190 | 10.605277 | ... | 11.111910 | 6.889096 | 10.636753 | 6.656603 | 11.054070 | 6.914937 | 8.949687 | 8.982860 | GSE53622 | cancer |
| GSM1296957 | 13.555346 | 4.772572 | 14.153843 | 4.388201 | 5.412374 | 9.339831 | 13.789576 | 4.211175 | 11.242888 | 10.518348 | ... | 10.998240 | 8.220715 | 10.645032 | 5.799432 | 10.951782 | 5.358962 | 8.951818 | 8.147058 | GSE53622 | normal |
| GSM1296958 | 13.396705 | 4.804828 | 13.948490 | 4.395992 | 5.627752 | 7.867446 | 13.424588 | 4.097212 | 10.568927 | 10.666406 | ... | 10.498048 | 8.145627 | 11.452488 | 6.164146 | 11.492929 | 6.189310 | 9.091511 | 10.021106 | GSE53622 | cancer |
| GSM1296959 | 13.843843 | 4.563550 | 14.390648 | 4.697154 | 5.511075 | 8.943584 | 14.181927 | 4.766994 | 10.418466 | 10.924152 | ... | 10.680012 | 8.450327 | 10.966135 | 6.482977 | 10.869259 | 6.683605 | 9.321499 | 9.278717 | GSE53622 | normal |
| GSM1296960 | 13.505687 | 4.750858 | 14.049400 | 4.476174 | 5.753380 | 8.475744 | 14.255647 | 4.344796 | 10.189663 | 10.651861 | ... | 9.778142 | 7.615217 | 10.570247 | 5.861632 | 11.168351 | 6.343246 | 8.793520 | 11.562505 | GSE53622 | cancer |
5 rows × 71586 columns
# We remove the dataset and sample type columns from the data frame and
# store the data frame in X
x = rnadataset.drop(labels=["dataset", "sample type"], axis=1)
# We store the dataset and sample type columns in the labels data frame
labels = rnadataset.loc[:, ["dataset", "sample type"]]
# Finding the top two principal components of the data
pca_decomposer = PCA(n_components=2)
pca_decomposer.fit(x)
PCA(n_components=2)In a Jupyter environment, please rerun this cell to show the HTML representation or trust the notebook.
PCA(n_components=2)
import seaborn as sns
x_reduced = pca_decomposer.transform(x)
# Display a scatterplot of the transformed dataset
plt.figure(figsize=(8, 6))
plt.title("PCA plot", size=24)
plt.xlabel("PC1", size=16)
plt.ylabel("PC2", size=16)
sns.scatterplot(x=x_reduced[:, 0], y=x_reduced[:, 1], hue=labels["sample type"]);
Даже не понимая, что за признаки у нас в колонках, мы видим несколько интересных вещей.
Во-первых, синяя точка на оранжевой территории и оранжевая на синей — видимо, выбросы.
Кроме этого мы видим, что почему-то у нас есть 4 почти равноудаленных кластера, по два на рак и нормальную ткань
# Run PCA on the features
x_reduced = pca_decomposer.transform(x)
# Display a scatterplot of the transformed dataset
plt.figure(figsize=(8, 6))
plt.title("PCA plot", size=24)
plt.xlabel("PC1", size=16)
plt.ylabel("PC2", size=16)
sns.scatterplot(
x=x_reduced[:, 0],
y=x_reduced[:, 1],
hue=labels["sample type"],
style=labels["dataset"],
);
Видим, что в пределах данных одного эксперимента рак и опухоль разделяются хорошо. Но при этом данные между разными экспериментами отличаются так же, как и рак от опухоли.
Этот эффект называется батч-эффектом и говорит о том, что нужно нормализовывать данные в пределах эксперимента каким-то хитрым образом, чтобы научить модель на новых данных отличать рак от нормальной ткани.
Таким образом, с помощью PCA мы нашли выбросы и артефакт в данных.
Интересное направление в данных может не совпадать с направлением максимальной дисперсии.
Рассмотрим случай выборки, которая сгенерирована из двух вытянутых нормальных распределений, чьи основные оси неортогональны друг другу:
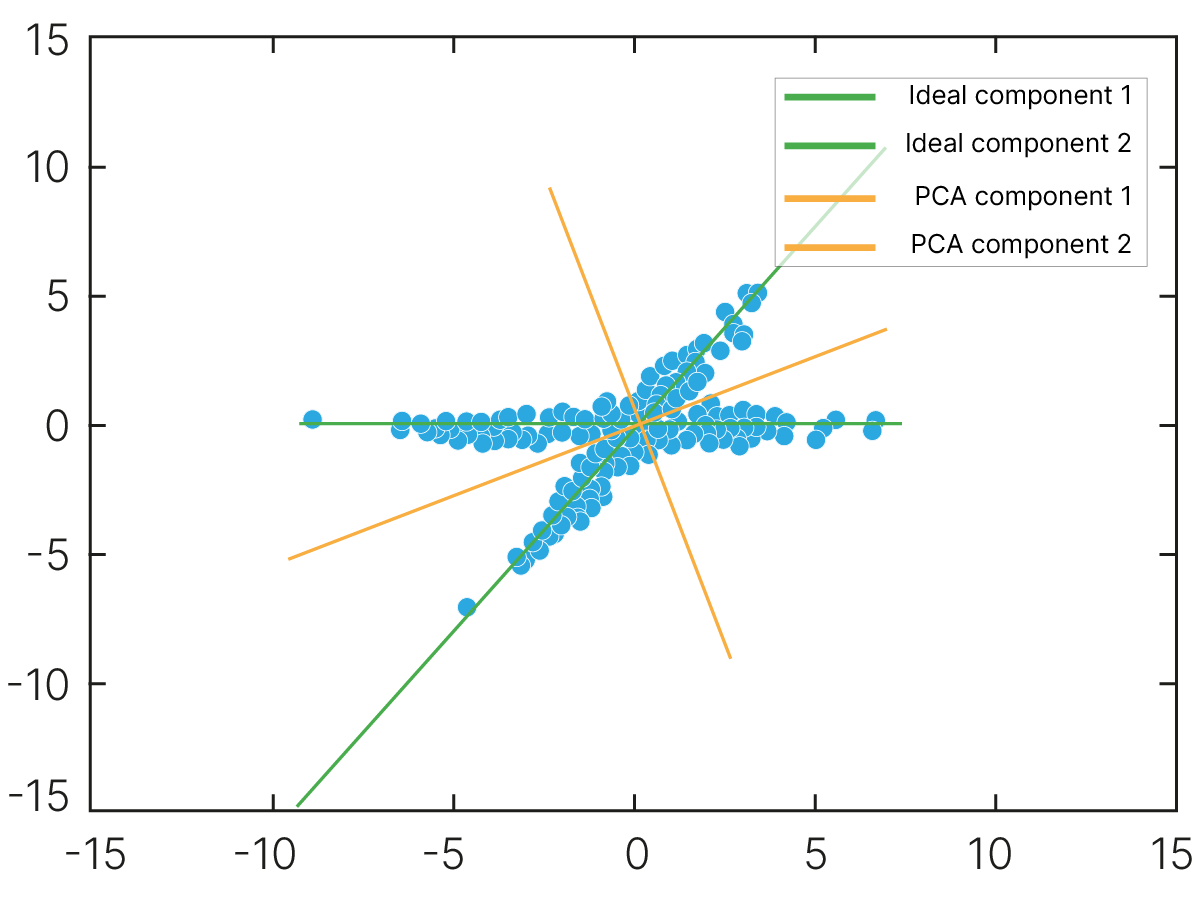
Выбранные оси могут не подходить для нашей задачи
В примере ниже дисперсии не отражают интересующие нас направления в данных:
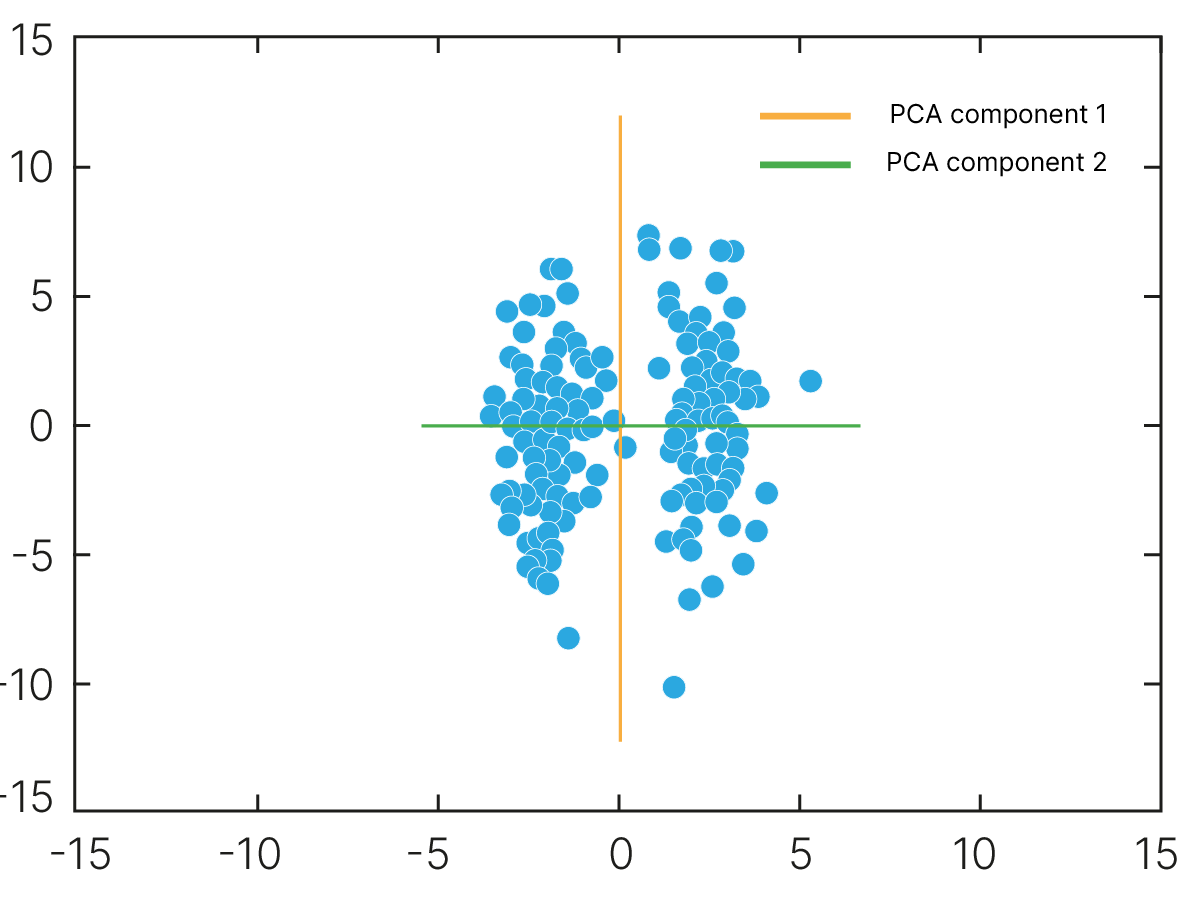
В данном случае метод главных компонент будет считать вертикальную компоненту более значимой для описания набора данных, чем горизонтальную.
Но, например, в случае, когда данные из левого и правого кластера относятся к разным классам, для их линейной разделимости вертикальная компонента является шумовой. Несмотря на это, метод главных компонент никогда ее шумовой не признает, и есть вероятность, что отбор признаков с его помощью выкинет из ваших данных значимые для решаемой вами задачи компоненты просто потому, что вдоль них значения имеют низкую дисперсию.
Справляться с такими ситуациями могут некоторые другие методы уменьшения размерности данных, например, метод независимых компонент (Independent Component Analysis, ICA).
Также бывают ситуации, когда оптимально спроецировать не на некоторую плоскость, а на многообразие (кривая плоскость), как показано на картинке ниже.
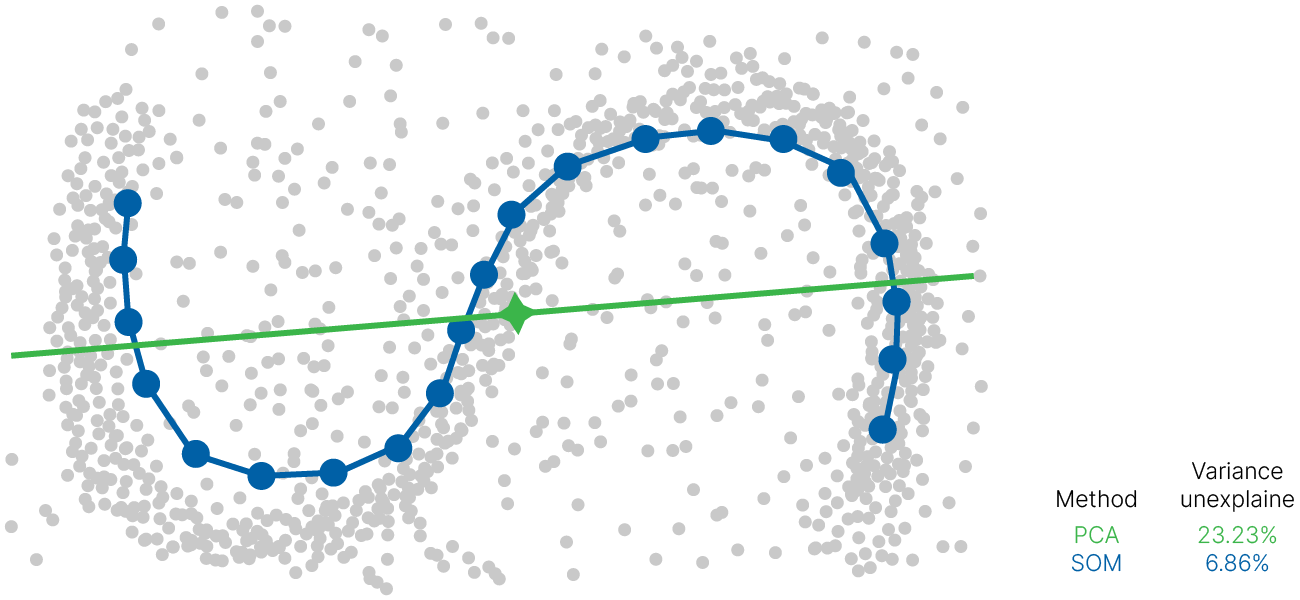
В данном случае оптимально спроецировать на S-образную кривую.
Как уже упоминалось, иногда невозможно захватить всю информацию линейной проекцией, хотя кривая поверхность с такой же размерностью это позволяет сделать. Одним из подходов к решению данной проблемы является задача перевода признаков в нелинейное пространство.
Kernel Trick избегает явного перевода наших признаков в пространство новых признаков, ведь пространства бывают очень большие, а нам бы хотелось сэкономить память компьютера. Оказывается, для PCA не важны собственно признаки объектов, а важны скалярные произведения между объектами.
И это скалярное произведением позволяет подсчитывать напрямую функция $k(\mathbf {x} ,\mathbf {x'})$, которую часто называют ядром или ядерной функцией (kernel, kernel function)
Бывают разные ядра, которые считают скалярное произведение в разных пространствах
Рассмотрим на примере:
# https://scikit-learn.org/stable/auto_examples/decomposition/plot_kernel_pca.html
from sklearn.datasets import make_circles
np.random.seed(42)
x, y = make_circles(n_samples=400, factor=0.3, noise=0.05, random_state=42)
Возьмем две концентрические окружности:
plt.figure(figsize=(5, 5))
plt.title("Original space")
class_0 = y == 0
class_1 = y == 1
sns.scatterplot(x=x[class_0, 0], y=x[class_0, 1], color="orange", s=30, label="Class 0")
sns.scatterplot(
x=x[class_1, 0], y=x[class_1, 1], color="#27a9e1", s=30, label="Class 1"
)
plt.xlabel("$x_1$")
plt.ylabel("$x_2$")
plt.legend()
plt.show()
Обычный PCA не может их разделить:
pca = PCA()
x_pca = pca.fit_transform(x)
plt.figure(figsize=(5, 5))
sns.scatterplot(
x=x_pca[class_0, 0], y=x_pca[class_0, 1], color="orange", s=30, label="Class 0"
)
sns.scatterplot(
x=x_pca[class_1, 0], y=x_pca[class_1, 1], color="#27a9e1", s=30, label="Class 1"
)
plt.title("Projection by PCA")
plt.xlabel("1st principal component")
plt.ylabel("2nd component")
plt.show()
А вот Kernel PCA справляется:
from sklearn.decomposition import KernelPCA
kpca = KernelPCA(kernel="rbf", fit_inverse_transform=True, gamma=10)
x_kpca = kpca.fit_transform(x)
plt.figure(figsize=(5, 5))
sns.scatterplot(
x=x_kpca[class_0, 0], y=x_kpca[class_0, 1], color="orange", s=30, label="Class 0"
)
sns.scatterplot(
x=x_kpca[class_1, 0], y=x_kpca[class_1, 1], color="#27a9e1", s=30, label="Class 1"
)
plt.title("Projection by KPCA")
plt.xlabel(r"1st principal component in space induced by $\phi$")
plt.ylabel("2nd component")
plt.show()
Хотя, конечно, и восстанавливать он будет не идеально: работал-то он в пространстве бОльшей размерности и оси строил там.
# 1. The 'kpca' variable is a KernelPCA object that is initialized with 'n_components' set to 2.
# 2. Then it applies the kernel function specified in the 'kernel' variable and then transforms the data based on the kernel, and gets the transformed data.
# 3. Then it returns the transformed data.
# 4. Then we get the inverse transformation by simply calling "kpca.inverse_transform(x_kpca)"
# 5. Finally, we plot the transformed data.
x_back = kpca.inverse_transform(x_kpca)
plt.figure(figsize=(5, 5))
sns.scatterplot(
x=x_back[class_0, 0], y=x_back[class_0, 1], color="orange", s=30, label="Class 0"
)
sns.scatterplot(
x=x_back[class_1, 0], y=x_back[class_1, 1], color="#27a9e1", s=30, label="Class 1"
)
plt.title("Original space after inverse transform")
plt.xlabel("$x_1$")
plt.ylabel("$x_2$")
plt.tight_layout()
plt.show()
Kernel PCA довольно чувствителен к выбору ядра.
К примеру, для данных, расположенных на трех окружностях:
в зависимости от выбора ядра мы будем получать совершенно разные отображение в спрямляющее пространство:
В связи с вышеописанными случаями, ниже мы рассмотрим более сильные методы.
Существуют и другие методы понижения размерности в данных. К примеру, t-SNE и UMAP.
Они немного иначе решают поставленную задачу: из пространства новой размерности мы не должны легко переходить в прежнее пространство. Взамен этого требуется, чтобы сохранялись расстояния между объектами. Причем, особое внимание уделяется расстояниям между близкими объектами. При этом расстояния между далекими объектами могут не сохраняться.
Идея состоит в том, чтобы не напрямую максимизировать дисперсию, а найти такое пространство, в котором расстояние между объектами будет сохраняться или по крайне мере не сильно меняться.
При этом будем больше беспокоиться о расстоянии между близкими объектами, нежели о расстоянии между далекими.
Рассмотрим алгоритм пошагово:
Шаг 1: Вычисляем матрицу попарных сходств объектов в пространстве исходной размерности. Затем преобразуем попарные сходства в условные вероятности.
Получаем, что объекты, считающиеся близкими в пространстве высокой размерности, имеют более высокую условную вероятность быть соседями и в пространстве низкой размерности.
Шаг 2: Иициализируем начальное положение объектов в пространстве низкой размерности. Так как мы сгенирировали его случайно, видим, что они сильно отличаются.
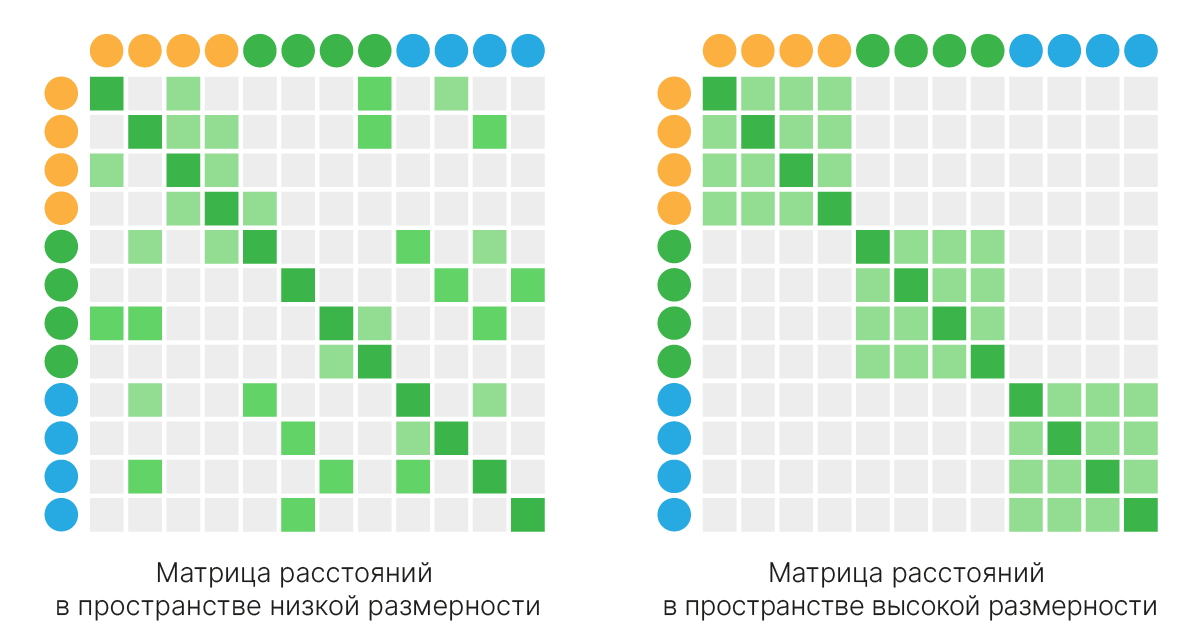
Шаг 3: Оптимизация. Осталось только привести сгенерированную матрицу к исходной. Делаем это с помощью градиентного спуска, минимизируя функцию потерь дивергенцию Кульбака — Лейблера 📚[wiki]:
$$KL(P||Q) = \sum_{i \neq j}{p_{ij} \log \frac{p_{ij}}{q_{ij}}},$$где:
$p_{ij}$ — условная вероятность близости между объектами $i$ и $j$ в пространстве исходной размерности,
$q_{ij}$ — условная вероятность близости между объектами $i$ и $j$ в пространстве низкой размерности.
В итоге мы хотим получить такое представление, чтобы объекты, которые находились близко в исходном пространстве, наиболее вероятно находились близко и в новом представлении.
Физическая аналогия:
Представьте себе комнату, в которой в воздухе висят шарики (наши объекты), которые соединены недеформированными пружинками, и время заморожено. Мы хотим из 3-мерного пространства (шарики в объеме комнаты), сделать представление в 2-мерном (шарики лежат на полу).
Если разморозить время, шарики упадут на пол. Тогда пружинки начнут стягивать шарики, которые были близки, и отталкивать шарики, которые упали близко, но были далеки. После того, как все шарики прекратят движение по полу и система из связанных шариков окажется в состоянии покоя, распределение расстояний между шариками на плоскости будет приближением для исходного распределения расстояний в объеме комнаты. Эта физическая аналогия показывает то, что происходит при минимизации дивергенции Кульбака — Лейблера.
Можем увидеть такой эффект на демонстрации:
# source: https://bost.ocks.org/mike/
from IPython.display import HTML
!wget -qN https://edunet.kea.su/repo/EduNet-web_dependencies/dev-2.0/L04/tSNE.html
HTML(filename="./tSNE.html")
Мы будем применять t-SNE не на исходных данных, а на 9-ти первых компонентах PCA. Для того, чтобы t-SNE сходился лучше, в качестве изначальных координат точек в новом пространстве можно использовать не случайный шум, а первые две компоненты PCA.
Преследуем две цели: уменьшить время работы t-SNE (который работает очень медленно) и убрать эффект шума на t-SNE — он может на него реагировать, особенно при условии, что схождения к минимуму мы можем не дождаться.
from sklearn import manifold
# 1. Firstly we reduce the dimensionality of the data to 6 features using PCA.
# 2. Then we take the first two PCA components and use this
# as an initial approximation for the T-SNE algorithm.
# 3. Then we fit T-SNE on the data and plot the first two dimensions
# of the T-SNE output, which are represented in green.
# 4. The visualization makes clear that there are distinct clusters in our data
scRNAseq = pd.read_csv(
"https://edunet.kea.su/repo/EduNet-web_dependencies/datasets/scRNAseq_CITEseq.txt",
sep="\t",
)
x = scRNAseq.iloc[:, 0:-1] # features
y = scRNAseq.iloc[:, -1] # labels
# taking the log of the data.
x = np.log(x + 1)
x_reduced = PCA(n_components=9).fit_transform(x)
model = manifold.TSNE(
n_components=2,
init=x_reduced[:, 0:2], # often use as a reasonable approximation
perplexity=40, # important parameter
verbose=2,
learning_rate="auto",
)
manifold = model.fit_transform(x_reduced)
plt.figure(figsize=(10, 5))
plt.scatter(manifold[:, 0], manifold[:, 1], c=y, cmap="tab20", s=20)
plt.title("TSNE: scRNAseq", fontsize=25)
plt.xlabel("TSNE1", fontsize=22)
plt.ylabel("TSNE2", fontsize=22)
plt.show()
[t-SNE] Computing 121 nearest neighbors... [t-SNE] Indexed 8617 samples in 0.030s... [t-SNE] Computed neighbors for 8617 samples in 0.988s... [t-SNE] Computed conditional probabilities for sample 1000 / 8617 [t-SNE] Computed conditional probabilities for sample 2000 / 8617 [t-SNE] Computed conditional probabilities for sample 3000 / 8617 [t-SNE] Computed conditional probabilities for sample 4000 / 8617 [t-SNE] Computed conditional probabilities for sample 5000 / 8617 [t-SNE] Computed conditional probabilities for sample 6000 / 8617 [t-SNE] Computed conditional probabilities for sample 7000 / 8617 [t-SNE] Computed conditional probabilities for sample 8000 / 8617 [t-SNE] Computed conditional probabilities for sample 8617 / 8617 [t-SNE] Mean sigma: 0.412418 [t-SNE] Computed conditional probabilities in 0.315s [t-SNE] Iteration 50: error = 73.6012802, gradient norm = 0.0086984 (50 iterations in 6.458s) [t-SNE] Iteration 100: error = 73.2053909, gradient norm = 0.0022528 (50 iterations in 6.333s) [t-SNE] Iteration 150: error = 73.0697556, gradient norm = 0.0015815 (50 iterations in 5.498s) [t-SNE] Iteration 200: error = 72.9788666, gradient norm = 0.0012755 (50 iterations in 6.135s) [t-SNE] Iteration 250: error = 72.9088440, gradient norm = 0.0010994 (50 iterations in 5.282s) [t-SNE] KL divergence after 250 iterations with early exaggeration: 72.908844 [t-SNE] Iteration 300: error = 2.3898752, gradient norm = 0.0103519 (50 iterations in 6.397s) [t-SNE] Iteration 350: error = 2.0336800, gradient norm = 0.0097805 (50 iterations in 4.792s) [t-SNE] Iteration 400: error = 1.8593026, gradient norm = 0.0087867 (50 iterations in 4.252s) [t-SNE] Iteration 450: error = 1.7605443, gradient norm = 0.0078783 (50 iterations in 4.721s) [t-SNE] Iteration 500: error = 1.6970280, gradient norm = 0.0072891 (50 iterations in 3.847s) [t-SNE] Iteration 550: error = 1.6515671, gradient norm = 0.0068512 (50 iterations in 3.881s) [t-SNE] Iteration 600: error = 1.6176370, gradient norm = 0.0063860 (50 iterations in 4.277s) [t-SNE] Iteration 650: error = 1.5913348, gradient norm = 0.0061006 (50 iterations in 3.671s) [t-SNE] Iteration 700: error = 1.5707281, gradient norm = 0.0056225 (50 iterations in 3.646s) [t-SNE] Iteration 750: error = 1.5543432, gradient norm = 0.0052370 (50 iterations in 4.297s) [t-SNE] Iteration 800: error = 1.5411797, gradient norm = 0.0047180 (50 iterations in 3.635s) [t-SNE] Iteration 850: error = 1.5311689, gradient norm = 0.0041818 (50 iterations in 3.612s) [t-SNE] Iteration 900: error = 1.5237211, gradient norm = 0.0034336 (50 iterations in 4.280s) [t-SNE] Iteration 950: error = 1.5180955, gradient norm = 0.0027117 (50 iterations in 3.573s) [t-SNE] Iteration 1000: error = 1.5136478, gradient norm = 0.0025790 (50 iterations in 3.532s) [t-SNE] KL divergence after 1000 iterations: 1.513648
И покрасим по разметке, которая нам известна из эксперимента. Видим, что разделение очень хорошее.
perplexity — чем больше perplexity, тем более глобально мы смотрим на структуру.
metric — как считаются расстояния между точками — metric. По умолчанию используется евклидово расстояние, но часто помогают и другие (например, косинусное).
learning_rate — шаг градиентного спуска, тоже влияет на полученное представление.
Низкоразмерное представление, которое вы получите, будет отличаться между запусками, если не зафиксировать random seed. Может отличаться довольно сильно.
Если у вас появились новые данные, то добавить их на представление, полученное при помощи t-SNE ранее — нетривиальная задача. Для разных областей есть свои "подгоны", но все это эвристика.
Расстояния между кластерами точек могут ничего не значить (плохо сохраняются далекие расстояния):

Размеры кластеров ничего не значат и могут сильно отличаться от исходных:
Можно увидеть не ту структуру, которая по идее должна быть:
Из-за указанных недостатков результат t-SNE нужно с осторожностью использовать для кластеризации.
[video] 📺 StatQuest: t-SNE, Clearly Explained
[article] 🎓 The art of using t-SNE for single-cell transcriptomics — cтатья по применению t-SNE в биологии
UMAP — uniform manifold approximation and projection.
[video] 📺 scRNA-seq: Dimension reduction (PCA, tSNE, UMAP)
Использует похожую на t-SNE идею, но иначе, в результате чего получает много выгодных бонусов.
Внутри себя метод строит граф, в котором ребрами соединены между собой $k$ ближайших соседей. При этом эти ребра неравноправны: если для данной пары точек расстояние между ними сильно больше, чем расстояния между ними и другими точками, то и ребро будет иметь маленький вес.

Далее задача состоит в том, чтобы в пространстве более низкой размерности получился граф, похожий на тот, который был в пространстве высокой размерности. Для этого оптимизируем низкоразмерное представление градиентным спуском.
from IPython.display import clear_output
!pip install -q umap-learn
!pip install -q --upgrade tbb
clear_output()
from umap import UMAP
# Converts the original expression matrix (scRNAseq) into a 9-dimensional PCA space
x_reduced = PCA(n_components=9).fit_transform(x) # x_scRNAseq
# Initializes UMAP with the PCA components
model = UMAP(
n_components=2,
min_dist=1,
n_neighbors=93,
init=x_reduced[:, 0:2],
# it is recommended to use the first two components of PCA for initialization of UMAP and t-SNE
n_epochs=1000,
verbose=2,
)
# Runs the UMAP algorithm on the PCA transformed data
umap = model.fit_transform(x_reduced)
clear_output()
# Plots the results of the UMAP transformation
plt.figure(figsize=(10, 5))
plt.scatter(umap[:, 0], umap[:, 1], c=y, cmap="tab20", s=20)
plt.title("UMAP: scRNAseq", fontsize=25)
plt.xlabel("UMAP1", fontsize=22)
plt.ylabel("UMAP2", fontsize=22)
plt.show()
n_neighbors — число соседей, которые ищутся для каждой точки. Влияет на то, насколько глобально мы смотрим на структуру данных.
min_dist — влияет на то, насколько близко могут находиться между собой точки в новом представлении.
metric — определяет, как считаются расстояния между точками. По умолчанию Евклидово расстояние, но это не всегда дает лучший результат.
Выдает результаты, похожие на t-SNE, но работает в разы быстрее
UMAP может проецировать точки из новых датасетов на уже имеющееся представление.
Если у вас есть признаки, сильно отличающиеся по своим свойствам, то можно построить для них представления отдельно с разными метриками, а далее объединить их.
Параметры n_neighbours и min_dist намного понятнее их аналога — perplexity.
Кроме того, его можно использовать для понижения размерности не только до 2-3 (в целях визуализации), но и для больших размерностей, с которыми потом работать другими методами (хотя здесь надо быть аккуратным, он тоже склонен деформировать дальние расстояния)
Плохая идея — интерпретировать расстояния между кластерами и их размеры для 2D представлений.
Но в случае UMAP это менее выражено.
Если же отображать в пространство размерности большей, чем два, то можно получить и очень хорошие представления. Но это все эвристика, может и не повезти.
UMAP позволяет передать в него метки объектов — вы можете получать представление, которое оптимизировано под имеющуюся у вас информацию о кластерах.
UMAP позволяет передать в него метки только для части объектов — вы можете получать представление, которое оптимизировано под имеющуюся у вас информацию о кластерах, но при этом не оставляет без внимания объекты без меток.
Методы понижения размерности, такие как UMAP и t-SNE, могут быть использованы как эффективный метод предобработки данных для последующей передачи в алгоритмы кластеризации на основе плотности 📚[wiki]. Данные методы понижения размерности не полностью сохраняют информацию о плотности распределения объектов при осуществлении проекции в низкоразмерное пространство, поэтому следует относиться к такой предобработке с некоторой осторожностью (рекомендуем ознакомиться с Clustering on the output of t-SNE ✏️[blog] на stackexchange). Несмотря на эти опасения, все же есть веские причины для использования данных методов понижения размерности в качестве этапа предварительной обработки для кластеризации. Как и при любом подходе к кластеризации, необходимо провести исследование и оценку полученных кластеров, чтобы попытаться валидировать разбиение на них, если это возможно.
Загрузим датасет с одноканальными $8 \times 8$ изображениями рукописных цифр:
from sklearn import datasets
digits = datasets.load_digits(n_class=10)
x = digits.data
y = digits.target
n_samples, n_features = x.shape
print(x.shape)
(1797, 64)
Понизим размерность данных $64 \rightarrow 2$ при помощи t-SNE:
from sklearn import manifold
# t-SNE embedding of the digits dataset
tsne = manifold.TSNE(n_components=2, init="pca", random_state=42, learning_rate="auto")
x_tsne = tsne.fit_transform(x)
Понизим размерность данных $64 \rightarrow 2$ при помощи UMAP:
umap = UMAP(n_neighbors=5)
x_umap = umap.fit_transform(x)
Визуализируем результаты:
from matplotlib import offsetbox
def plot_embedding(x, title=None):
x_min, x_max = np.min(x, 0), np.max(x, 0)
x = (x - x_min) / (x_max - x_min) # normalization of x to (0..1) range
plt.figure()
ax = plt.subplot(111)
for i in range(x.shape[0]):
plt.text(
x[i, 0],
x[i, 1],
str(y[i]),
color=plt.cm.Set1(y[i] / 10.0),
fontdict={"weight": "bold", "size": 9},
)
if hasattr(offsetbox, "AnnotationBbox"):
# only print thumbnails with matplotlib > 1.0
shown_imgs = np.array([[1.0, 1.0]]) # just something big
for i in range(x.shape[0]):
dist = np.sum((x[i] - shown_imgs) ** 2, 1)
if np.min(dist) < 4e-3:
# don't show points that are too close
continue
shown_imgs = np.r_[shown_imgs, [x[i]]]
img_box = offsetbox.AnnotationBbox(
offsetbox.OffsetImage(digits.images[i], cmap=plt.cm.gray_r), x[i]
)
ax.add_artist(img_box)
plt.xticks([]), plt.yticks([])
if title is not None:
plt.title(title)
plt.figure()
ax = plt.subplot(111)
for i in range(x.shape[0]):
plt.text(
x[i, 0],
x[i, 1],
str(y[i]),
color=plt.cm.Set1(y[i] / 10.0),
fontdict={"weight": "bold", "size": 9},
)
plt.xticks([]), plt.yticks([])
if title is not None:
plt.title(title)
# t-SNE embedding of the digits dataset
plot_embedding(x_tsne, "t-SNE embedding of the digits")
# UMAP embedding of the digits
plot_embedding(x_umap, "UMAP embedding of the digits")
Как нетрудно заметить, при использовании UMAP точки из датасета при проекции в низкоразмерное пространство оказались расположены более "кучно", нежели чем при использовании t-SNE.
import sklearn.cluster as cluster
kmeans_labels_on_raw = cluster.KMeans(n_clusters=10, n_init="auto").fit_predict(x)
from sklearn.metrics import adjusted_rand_score, adjusted_mutual_info_score
def plot_clustering_metrics(true_l, pled_l, title):
ari = adjusted_rand_score(true_l, pled_l)
ami = adjusted_mutual_info_score(true_l, pled_l)
fig, ax = plt.subplots(1, 1, figsize=(10, 6))
plt.title(f"Clustering metrics for {title}\n\n 1.0 is best")
width = 0.75
ind = np.arange(2)
ax.barh(ind, [ari, ami], width)
ax.grid(axis="x")
ax.set_xlim([0, 1.0])
for i, v in enumerate([ari, ami]):
ax.text(v + 0.01, i, f"{v:1.2f}", color="black")
ax.set_yticks(ind)
ax.set_yticklabels(["ARI", "AMI"], minor=False)
plt.show()
plot_clustering_metrics(y, kmeans_labels_on_raw, "kNN on raw dataset")
Теперь передадим K-Means данные после понижения размерности:
kmeans_labels_on_x_tsne = cluster.KMeans(n_clusters=10, n_init="auto").fit_predict(
x_tsne
)
kmeans_labels_on_x_umap = cluster.KMeans(n_clusters=10, n_init="auto").fit_predict(
x_umap
)
plot_clustering_metrics(y, kmeans_labels_on_x_tsne, "kNN on t-SNE data")
plot_clustering_metrics(y, kmeans_labels_on_x_umap, "kNN on UMAP data")
Применяя методы понижения размерности на этапе предобработки данных, нам удалось значительно улучшить качество кластеризации. Использование UMAP в разобранном нами примере позволило достичь лучших результатов по сравнению с t-SNE и получить прирост практически в 20% относительно метрик качества кластеризации.
Литература
Проблемы при работе с реальными данными:
Кластеризация:
Кодирование признаков:
Разведочный анализ данных:
Генерация признаков:
Отбор признаков:
Задача понижения размерности:
[article] 🎓 The art of using t-SNE for single-cell transcriptomics
Дополнительно: